Notre étude fournit des preuves de la présence du SRAS-CoV-2 dans le système vasculaire coronarien humain et démontre un tropisme viral pour les macrophages des lésions vasculaires chez les personnes atteintes de COVID-19 sévère. Nous avons trouvé des preuves de réplication du SRAS-CoV-2 dans tous les coronaires d’autopsie humaine analysés, quelle que soit leur classification pathologique, bien que la réplication virale ait été la plus élevée dans les lésions coronariennes PIT — les lésions précoces qui progressent vers des plaques athérosclérotiques plus avancées11,12. Il est bien établi que, à mesure que les lésions progressent vers des stades plus avancés, tels que les fibroathéromes et les plaques fibrocalciques, les macrophages et les cellules moussantes deviennent dysfonctionnels en raison d’une exposition prolongée aux lipides et d’un microenvironnement inflammatoire11,12. Dans les lésions PIT précoces, les macrophages qui tentent d’éliminer les lipides inflammatoires (par exemple, les oxLDL) accumulent un excès de cholestérol et de triglycérides dans les gouttelettes lipidiques cytoplasmiques12 peut être plus sujet aux infections virales. Le SRAS-CoV-2 a montré un tropisme plus fort pour les lésions artérielles que la graisse périvasculaire correspondante, qui était liée au degré d’infiltration des macrophages, compatible avec la réplication virale plus élevée dans les lésions PIT et les fibroathéromes, où les macrophages sont plus répandus. D’autres ont déjà signalé la présence d’ARNv du SRAS-CoV-2 dans le cœur et l’aorte ainsi que dans d’autres organes distants8,14,59,60. Nos données démontrent de manière concluante que le SRAS-CoV-2 est capable d’infecter et de reproduire dans les macrophages dans le vasculature coronaire des patients atteints de COVID-19. De plus, le SRAS-CoV-2 se reproduit préférentiellement dans les cellules en mousse par rapport à d’autres macrophages, en raison d’une réponse IFN réduite de type I qui conduit à l’ARNV et à l’accumulation de protéines virales, suggérant que ces cellules pourraient servir de réservoir de débris viraux du SRAS-CoV-2 dans la plaque athérosclérotique. Nos résultats démontrent que, bien que la réponse IFN de type I soit restée élevée dans les macrophages infectés par le SRAS-CoV-2, indiquant un état antiviral soutenu, les cellules en mousse infectées ne présentaient qu’une expression transitoire de l’expression du gène IFN de type I. Associé à l’expression réduite de gènes lysosomaux et à une réponse inflammatoire plus faible à l’infection par le SRAS-CoV-2,la réduction de l’IFN de type I peut contribuer à la persistance du SRAS-CoV-2 observée dans les spécimens d’autopsie athéroscléreuse analysés. Les macrophages résidant dans le tissu vasculaire peuvent subir l’auto-renouvellement, contribuant à leur entretien à long terme61, et, dans les études de transplantation cardiaque, il a été démontré que les macrophages cardiaques ont une demi-vie de 8,8 ans62. Par conséquent, il est possible que les macrophages agissent comme des réservoirs d’ARNv SRAS-CoV-2 dans des plaques athérosclérotiques. Nous avons également identifié des VSMC infectés dans des échantillons d’autopsie coronaire. Bien que le nombre de VSMC infectés soit inférieur à celui des macrophages infectés, les VSMC ont une demi-vie de 270 – 400 d63 et peut contribuer à la persistance du SRAS-CoV-2 dans la paroi artérielle.
- Article
- Accès libre
- Publié:
L’infection au SRAS-CoV-2 déclenche des réponses inflammatoires pro-athérogènes dans les vaisseaux coronariens humains
Recherche cardiovasculaire sur la nature volume 2, pages899 – 916 (2023)
Abstrait
Les patients atteints de la maladie à coronavirus 2019 (COVID-19) présentent un risque accru de complications cardiovasculaires ischémiques jusqu’à 1 an après l’infection. Bien que la réponse inflammatoire systémique à l’infection par le coronavirus 2 du syndrome respiratoire aigu sévère (SARS-CoV-2) contribue probablement à ce risque cardiovasculaire accru, on ignore si le SRAS-CoV-2 infecte directement le système vasculaire coronaire et les plaques athérosclérotiques associées. Ici, nous rapportons que l’ARN viral du SRAS-CoV-2 est détectable et se réplique dans les lésions coronaires prises à l’autopsie de cas graves de COVID-19. Le SRAS-CoV-2 ciblait les macrophages de la plaque et présentait un tropisme plus fort pour les lésions artérielles que la graisse périvasculaire adjacente, en corrélation avec les niveaux d’infiltration des macrophages. L’entrée du SRAS-CoV-2 a été augmentée dans les macrophages primaires chargés de cholestérol et dépendant, en partie, de la neuropiline-1.Le SRAS-CoV-2 a induit une réponse inflammatoire robuste dans les macrophages cultivés et les explants vasculaires athérosclérotiques humains avec sécrétion de cytokines connues pour déclencher des événements cardiovasculaires. Nos données établissent que le SRAS-CoV-2 infecte les vaisseaux coronaires, induisant une inflammation de la plaque qui pourrait déclencher des complications cardiovasculaires aiguës et augmenter le risque cardiovasculaire à long terme.
Principal
La maladie à coronavirus 2019 (COVID-19), causée par le coronavirus 2 du syndrome respiratoire aigu sévère (SARS-CoV-2), est uniquement marquée par un tropisme tissulaire extraordinaire et un éventail de présentations cliniques, de l’infection asymptomatique à la détresse respiratoire aiguë, à l’insuffisance multiviscérale et à la mort1. Événements cardiovasculaires ischémiques, tels que l’infarctus aigu du myocarde (AMI) et accident vasculaire cérébral, en raison de la perturbation sous-jacente d’une plaque athéroscléreuse enflammée de façon chronique2, sont établies les complications cliniques de COVID-19 (réfs. 1,3‘). L’AMI et l’accident vasculaire cérébral peuvent être déclenchés par plusieurs infections virales respiratoires aiguës, y compris le virus de la grippe4. Cependant, les patients atteints de COVID-19 sont > 7 fois plus susceptibles d’avoir un AVC que les patients atteints de grippe5, et leur risque d’AMI et d’AVC reste élevé jusqu’à 1 an après l’infection6. La réponse inflammatoire extrême qui se produit dans les cas graves de COVID-19, également connu sous le nom de tempête de cytokines7, contribue probablement à l’augmentation du risque d’AMI et d’AVC. Cependant, la possibilité que le SRAS-CoV-2 affecte directement le vasculature coronaire, comme documenté pour d’autres organes éloignés ( par exemple, rein, intestin, cerveau, tissu adipeux et myocarde )8, reste largement inexploré. Dans les poumons, les lésions tissulaires sont aggravées par une puissante activation inflammasomique dans les macrophages détectant le virus SRAS-CoV-29. Une réponse similaire dans les macrophages infiltrant les vaisseaux artériels affectés par le virus pourrait augmenter l’inflammation de la plaque et le risque d’AMI et d’accident vasculaire cérébral chez les patients atteints de COVID-19. Ici, nous montrons, dans des spécimens d’autopsie coronarienne de patients atteints de COVID-19, que les macrophages infiltrants ont été infectés par le SRAS-CoV-2. Les macrophages chargés de lipides (cellules de mousse), une caractéristique de l’athérosclérose à tous les stades de la maladie10, étaient plus sensibles à l’infection par le SRAS-CoV-2 que les autres macrophages, et cela dépendait du récepteur neuropiline-1 (NRP-1). Le SRAS-CoV-2 a induit une forte réponse inflammatoire pro-athérogène dans les macrophages et les cellules de mousse, qui a été largement récapitulée dans une infection ex vivo du SRAS-CoV-2 d’explants vasculaires humains. Cette réponse peut contribuer aux complications cardiovasculaires ischémiques chez les patients atteints de COVID-19.
L’IA spatiale détecte l’ARN du SRAS-CoV-2 dans les macrophages coronaires
Nous avons analysé les échantillons d’autopsie coronaire (n = 27 ) de huit patients avec RT – Diagnostic confirmé par PCR de COVID-19 entre mai 2020 et mai 2021. Des données démographiques et des caractéristiques cliniques, y compris des antécédents médicaux, des facteurs de risque cardiovasculaires et d’autres informations cliniques pertinentes, ont été obtenues auprès des patients ’ dossiers médicaux électroniques et rapports d’autopsie ( IRB i21-01587 ) ( Fig. 1a et tableaux supplémentaires 1 et 2). L’âge moyen était de 69,6 ans ( médiane, 71 ans; 59 – 84 ) et 75% des patients étaient des hommes ( 6/8 ). Les patients avaient une maladie coronarienne ( 8/8 ); trois facteurs de risque cardiovasculaire ou plus, tels que l’hypertension ( 8/8 ), le surpoids ou l’obésité ( 7/8 ), l’hyperlipidémie ( 7/8 ), diabète de type 2 ( 6/8 ) et maladie rénale chronique ( 4/8 ); et certains avaient des antécédents d’infarctus du myocarde ( 1/8 ) ou d’AVC ischémique ( 1/8 ) Fig. 1a et tableau supplémentaire 1). Une ischémie myocardique aiguë a été diagnostiquée lors de l’hospitalisation chez trois patients; un patient a développé un AVC; et quatre patients ont présenté une sténose coronarienne ( > 50% ) lors de l’autopsie. Les patients, à l’exception de celui qui a été déclaré mort avant l’admission, ont été hospitalisés pour une moyenne de 17,6 d (, 8 – 31 ) ( Tableau supplémentaire 2). Un patient ( Pt. 1 ) a été hospitalisé trois fois ( pour un total de 31 d ) après le premier diagnostic. Pt. 1 a développé AMI et est décédé lors de la troisième hospitalisation, environ 140 d après avoir été testé négatif pour COVID-19 ( Fig. 1a et tableau supplémentaire 2). La présentation clinique, le cours à l’hôpital de la maladie, les traitements COVID-19 ainsi que la pathologie d’autopsie macroscopique et microscopique obtenue à partir des dossiers médicaux de l’hôpital et des rapports d’autopsie sont résumés dans le tableau supplémentaire 2.
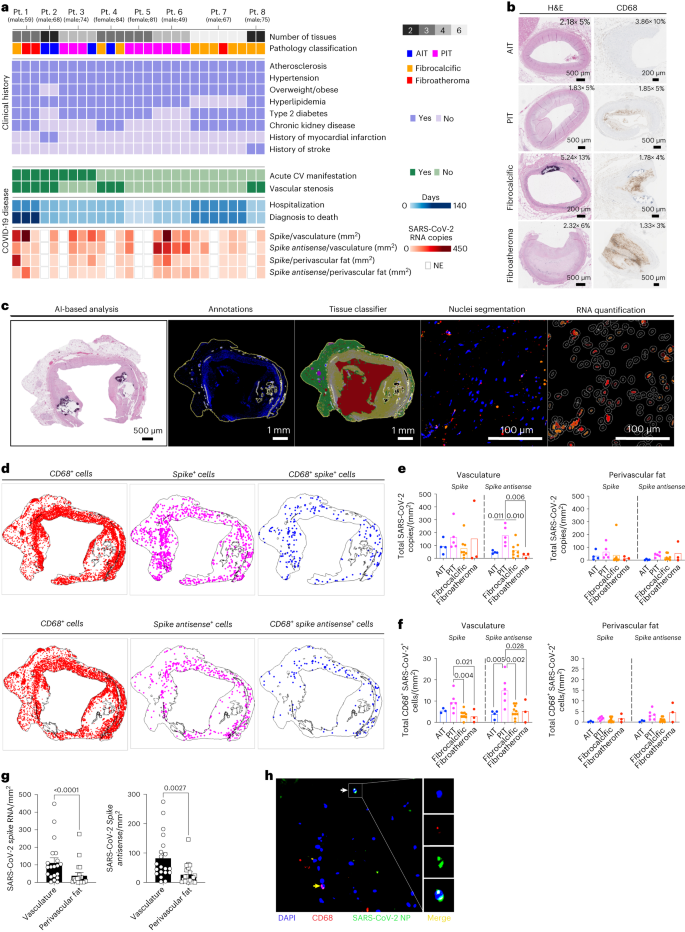
un, Carte thermique catégorique des échantillons d’autopsie coronaire (n = 27 ) de personnes décédées avec COVID-19 (n = 8 ) affiche leur classification du sexe, de l’âge et de la pathologie en AIT, PIT, plaques fibrocalcifiques et fibroathéromes. L’historique clinique de chaque patient est montré. Résumé des manifestations cardiovasculaires aiguës ( CV ) pendant la progression de la maladie COVID-19, sténose coronaire ( non: < 50%; oui: > 50% ), la durée de l’hospitalisation et le délai de décès après le diagnostic de COVID-19 sont également représentés. Numéros de copie d’ARN de l’ARNv antisens S et S normalisé en zone graisse vasculaire et périvasculaire ( mm2) sont affichés. NE, pas évalué. b, Images représentatives d’échantillons coronaires colorés avec coloration H&E et CD68 pour chaque classification pathologique. c, Images représentatives de l’analyse in situ basée sur l’IA RNA-FISH. Après des annotations semi-automatiques, un réseau neuronal basé sur l’IA a été utilisé pour classer le vasculature ( jaune ) et la graisse périvasculaire ( vert ). Le fond et les artefacts ( rouge ) ont été supprimés de l’analyse. Ensuite, l’analyse du classificateur de segmentation des noyaux et la quantification de l’ARN ont été effectuées en utilisant l’IA HALO et le flux de travail d’analyse spatiale. d, Images représentatives de l’analyse spatiale montrant l’emplacement de CD68 ARN, SRAS-CoV-2 S+ ou S antisense+ cellules et CD68+ Cellules à double positif à l’ARN SRAS-CoV-2. e, Diagrammes en barres montrant des copies totales de l’ARNv SARS-CoV-2 de l’antisens S et S normalisées par zone tissulaire (mm2) dans AIT (n = 4), FOSSE (n = 5), fibrocalciques (n = 8) et fibroathérome (n = 3) échantillons coronaires. f, Parcelles de barres montrant le total CD68+ SARS-CoV-2 S+ ou S antisens+ cellules dans les régions graisseuses vasculaires ou périvasculaires normalisées par zone tissulaire (mm2) dans AIT (n = 4), FOSSE (n = 5), fibrocalciques (n = 10) et fibroathérome (n = 3) échantillons coronaires. Une analyse statistique à sens unique de la variance (ANOVA) avec un test de Tukey’s post hoc pour des comparaisons multiples a été effectuée. g, SARS-CoV-2 S et S quantification antisens dans la graisse vasculaire et périvasculaire normalisée par la zone de tissu (mm2‘). Un test de rang signé de paires appariées Wilcoxon a été effectué (n = 20 par groupe). h, Images représentatives de SARS-CoV-2 NP, CD68 et fusionner dans les coronaires humains. La flèche blanche indique CD68+ SARS-CoV-2 NP+ la cellule et la flèche jaune indiquent CD68+ cellule. Pt., patient.
Des sections d’artères coronaires de toutes les autopsies ont été colorées avec de l’hématoxyline et de l’éosine ( H&E ) et classées par un pathologiste cardiovasculaire clinique ( N.N. ) comme épaississement adaptatif intimal ( AIT; n = 4 ), épaississement pathologique intimal avec infiltration de macrophages ( PIT; n = 10 ), plaque fibrocalcifique (n = 10 ) et fibroathérome (n = 3 ) ( Fig. 1a, b, Fig. D’AFFAIRES Extended. 1a, Fig. Supplémentaire. 1 et tableau supplémentaire 3) selon des critères établis11,12. Des caractéristiques pathologiques détaillées, y compris la présence de pool lipidique, de noyau nécrotique et d’inflammation adventielle, ont été notées (Données étendues Fig. 1a‘). Salissure immunohistochimique (IHC) pour CD68+ les cellules ont montré que CD68+ la numération cellulaire et les fréquences n’étaient pas significativement différentes chez les PIT, les lésions fibrocalcifiques et les fibroathéromes, alors qu’une plus grande surface colorée positive correspondant aux noyaux nécrotiques était observée chez les fibroathéromes (Fig. 1b, Données Extended Fig. 1b, c et Table Supplémentaire 3‘).
Identifier l’ARN viral SRAS-CoV-2 ( vRNA ) dans le vasculature coronaire autoptique des patients atteints de COVID-19, nous avons effectué une analyse par fluorescence d’ARN par hybridation in situ ( ARN-FISH ) pour l’ARNv codant pour la protéine de pointe ( S ). Pour établir si le SRAS-CoV-2 a infecté le vasculature coronaire humain, nous avons également sondé le brin antisens du gène S (S antisense), qui n’est produit que pendant la réplication virale. Une sonde CD68 a été utilisée pour identifier les macrophages infiltrant les vaisseaux coronaires dans les mêmes sections et établir la localisation cellulaire de l’ARNv SRS-CoV-2. Étant donné la capacité du SRAS-CoV-2 à infecter les dépôts graisseux et à accumuler de l’ARNV dans le tissu adipeux pour déclencher une forte réponse pro-inflammatoire13,14, nous avons utilisé une approche d’intelligence artificielle de réseau neuronal ( AI ) pour classer la paroi artérielle coronaire et la graisse périvasculaire dans chaque échantillon et segmentation des noyaux pour quantifier les sondes d’ARNscope dans les cellules infiltrant les deux tissus ( Fig. 1c, d). Dans la paroi artérielle coronaire, l’ARNv codant pour la protéine S et le brin antisens du gène S ont été détectés à différents degrés dans toutes les sections de tous les patients, indiquant la présence d’ARNV et l’activité réplicative du virus. Le numéro de copie du gène SRAS-CoV-2 S était similaire dans les lésions coronariennes de l’IA, du PIT, du fibrocalcifique et du fibroathérome ( Fig. 1e). Cependant, les coronaires PIT ont montré un nombre de copies significativement plus élevé du brin antisens du gène S (S antisense), indiquant une réplication virale plus élevée dans la paroi vasculaire de ces lésions (Fig. 1e‘). En particulier, CD68+ les cellules exprimant à la fois le SRAS-CoV-2 S et le brin antisens de S étaient significativement plus élevées dans le système vasculaire des coronaires PIT par rapport à d’autres pathologies (Fig. 1f‘). Dans la graisse périvasculaire, l’ARNv codant pour la protéine S a été détecté dans 20 des 21 sections, et l’antisens S a été détecté dans 19 des 21 sections (Fig. 1a‘). Dans l’ensemble, chaque patient a présenté au moins une section positive pour le VRNA antisens S et S (Fig. 1ae, f‘). Notamment, la quantité d’ARNv des brins antisens S et S était significativement plus faible dans la graisse périvasculaire que dans la paroi artérielle correspondante dans tous les échantillons (Fig. 1g‘). L’accumulation de protéines virales dans les coronaires a été confirmée par immunofluorescence (Fig. 1h‘).
Lésions artérielles PIT, qui semblaient plus sensibles à l’infection par le SRAS-CoV-2, contient significativement plus de cellules que les autres types de lésions et 4,8 fois plus de cellules que le tissu périvasculaire correspondant (2 691,8 ± 288,7 versus 697,6 ± 159,3 cellules par mm2; P < 0,0001) (Données étendues Fig. 1d‘). Le nombre d’ARN CD68+ les cellules du système vasculaire coronaire étaient significativement plus élevées dans les PIT et les fibroathéromes que dans les autres lésions (Données étendues Fig. 1e), ce qui correspond à un nombre plus élevé d’ARN CD68+ cellules dans la graisse périvasculaire des lésions PIT que les lésions fibrocalcifiques et similaires aux fibroathéromes (Données étendues Fig. 1e), suggérant un rôle contributif de l’infection directe des macrophages dans la sensibilité coronaire à l’infection par le SRAS-CoV-2. Cette possibilité a été suggérée par la fréquence significativement plus élevée du CD68+ cellules exprimant le brin antisens S de l’ARNv dans les lésions PIT et la graisse périvasculaire correspondante ( Données étendues Fig. 1f) ainsi que l’association importante des copies d’ARNv du SRAS-CoV-2 avec numéro de copie CD68 dans la paroi artérielle et la graisse périvasculaire ( Données étendues Fig. 1g). L’accumulation d’ARNv SRAS-CoV-2 codant pour la protéine S était plus élevée dans le tissu entier et dans la paroi coronaire chez les patients atteints de COVID-19 avec des manifestations cardiovasculaires aiguës ( Données étendues Fig. 1h).
Pour étudier l’extension potentielle de l’infection au SRAS-CoV-2 à d’autres cellules, nous nous sommes concentrés sur les cellules musculaires lisses vasculaires ( VSMC ), sur la base de la distribution spatiale de la plupart des SRAS-CoV-2+ cellules à l’intérieur de la paroi artérielle des coronaires, et nous avons effectué des analyses de NAc qui ont été guidées par des analyses des ensembles de données de séquençage d’ARN monocellulaire humain et murin ( scRNA-seq ). Une analyse, utilisant des données scRNA-seq de coronaires humains malades ( Expression génétique Omnibus ( GEO ): GSE131780)15, a révélé que les VSMC expriment des niveaux de ACTA2 par rapport aux cellules myéloïdes, alors que l’expression de CD68 était spécifique aux macrophages ( Fig. Donnée étendue. 2a‘). De plus, nous avons analysé les données scRNA-seq de cellules dérivées de muscles lisses (VSMC) marquées par lignage, isolées des arches aortiques athérosclérotiques d’ApoE−/− Tomate souris16. Cette analyse a révélé que les CMLV dans les lésions athérosclérotiques n’exprimaient pas de niveaux significatifs de Cd68 et a maintenu un niveau élevé de Acta2 expression, bien que son expression ait été réduite par rapport aux conditions non-athérosclérotiques (Données étendues Fig. 2b‘). Sur la base de ces résultats, nous avons utilisé ACTA2 sonde pour identifier les VSMC et les macrophages d’origine VSMC comme ACTA2+, et nous avons inclus des sondes pour le VRNA antisens S et S dans l’analyse pour identifier ACTA2+ cellules infectées. Notamment, cette analyse a identifié S+ACTA2+ cellules et antisens+ACTA2+ cellules dans les coronaires humains ( Données étendues Fig. 2c); cependant, la médiane de la fréquence des VSMC infectés par le SRAS-CoV-2 ( ~ 0,14% ) chez la coronaire humaine était ~ 8 fois inférieure à celle des macrophages infectés ( ~ 1,2% ). Ces résultats montrent que, bien que le SRAS-CoV-2 puisse infecter les VSMC, le nombre de VSMC infectés dans les coronaires humains était inférieur au nombre de macrophages infectés. Étudier plus avant l’infection par le SRAS-CoV-2 des VSMC et des VSMC chargés de lipides, qui sont associés à l’athérosclérose17,18,19,20, nous avons infecté les VSMC de l’aorte humaine primaire, ainsi que les VSMC chargés de complexes cyclodextrine – cholestérol ( Données étendues Fig. 2d), avec l’isolat SRAS-CoV-2 USA WA1 / 2020. Environ 18% des VSMC cultivés et 13% des VSMC chargés de cholestérol étaient S+, et la fréquence de S antisense+ACTA2+ les cellules, indiquant une réplication virale, étaient ~ 2,6% ( Données étendues Fig. 2d, e). Conjugués à nos résultats in vitro, qui indiquent que plus de 79% des macrophages et plus de 90% des cellules en mousse sont S +, ainsi que la découverte que plus de 40% des deux types de cellules sont S antisense +, ces résultats montrent que, bien que le SRAS-CoV-2 puisse infecter les VSMC, les macrophages sont infectés à un taux plus élevé.
SARS-CoV-2 infection des macrophages humains et des cellules de mousse
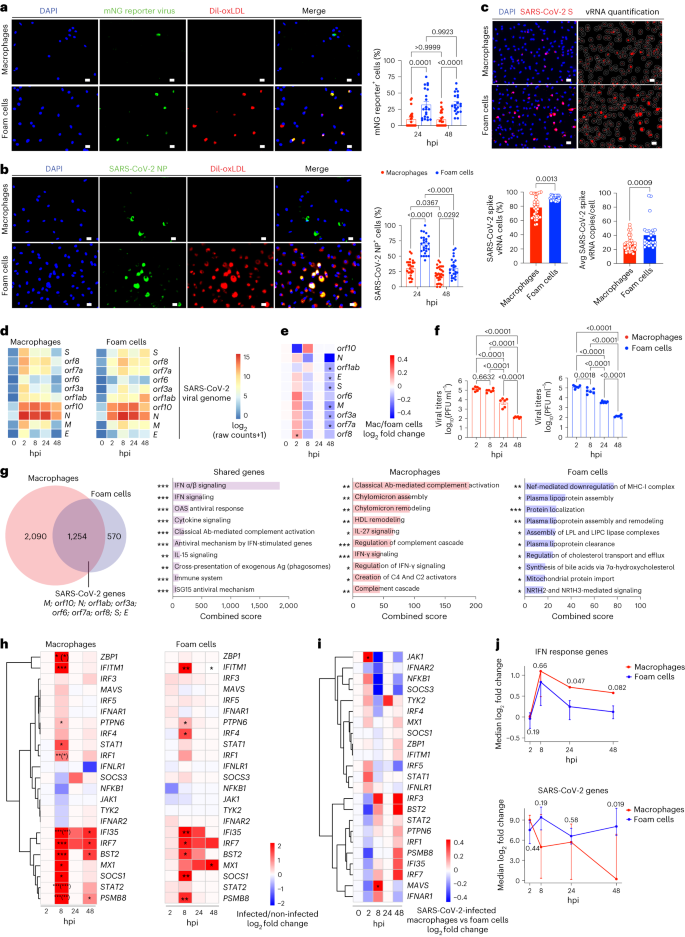
L’accumulation de macrophages chargés de cholestérol (cellules de mousse) est une caractéristique de l’athérosclérose à tous les stades de la maladie, de la PIT précoce aux lésions du fibroathérome tardif10,12. Étudier l’infection par le SRAS-CoV-2 des macrophages et des cellules de mousse, nous avons différencié les monocytes humains dérivés de cellules mononucléées du sang périphérique humain en macrophages et les avons traités avec des lipoprotéines de basse densité oxydées (oxLDL) complexées avec un colorant Dil (Dil-Ox-LDL) pour les différencier en cellules de mousse. Pour confirmer expérimentalement notre observation que le SRAS-CoV-2 peut infecter les macrophages de la plaque humaine, les macrophages et les cellules de mousse ont été infectés soit par le virus rapporteur icSARS-CoV-2 mNeonGreen (mNG), un virus modifié qui permet l’utilisation de la fluorescence mNG comme lecture de substitution pour la réplication virale21, ou avec l’isolat SARS-CoV-2 USA WA1/2020. l’expression de mNG a confirmé la capacité du SARS-CoV-2 à se répliquer dans les deux types de cellules, bien que la réplication soit plus élevée dans les cellules en mousse (Fig. 2a et Extended Data Fig. 3a‘). L’accumulation significativement plus élevée de nucléoprotéine (NP) dans les cellules en mousse par rapport aux macrophages infectés par l’isolat SARS-CoV-2 USA WA1/2020 (Fig. 2b et Extended Data Fig. 3b) a confirmé une susceptibilité plus élevée des cellules de mousse au virus. En fait, bien que la fréquence de NP+ les cellules de mousse ont diminué entre 24 heures après l’infection (hpi) et 48 hpi, la proportion de SARS-CoV-2 NP+ les cellules de mousse sont restées significativement plus élevées que celles du NP+ macrophages (Fig. 2b‘). Les cellules de mousse ont également accumulé plus d’ARNv S du SRAS-CoV-2 que les macrophages (Fig. 2c‘). le génome du vRNA du SRAS-CoV-2 était détectable dans les macrophages et les cellules de mousse dès 2 hpi, est resté élevé jusqu’à 24 hpi, mais a été réduit à 48 hpi dans les deux types de cellules (Fig. 2d et Extended Data Fig. 3d‘). Notamment, l’expression des gènes SARS-CoV-2 a diminué significativement plus rapidement dans les macrophages que dans les cellules en mousse, avec un vRNA significativement plus faible à 48 hpi (Fig. 2e). Cette décroissance temporelle dynamique des niveaux d’ARNV indique que les macrophages sont capables de nettoyer le virus plus rapidement que les cellules en mousse. Quoi qu’il en soit, bien qu’il soit sensible, les macrophages et les cellules en mousse n’ont pas soutenu une formation productive de particules virales infectieuses comme en témoignent les résultats du test de plaque à l’aide de cellules Vero E6 modifiées exprimant la transmembrane protéase sérine 2 et enzyme humaine de conversion de l’angiotensine 2 ( Vero E6-TMPRSS2-T2A-ACE2 ). Ces tests ont montré un déclin progressif des milieux conditionnés par le titre viral des macrophages infectés et des cellules en mousse ( Fig. 2f et Fig. Donnée étendue. 3c). Ces résultats sont cohérents avec l’infection abortive, un résultat courant de nombreuses infections virales, les cellules infectées ne produisant pas de virus compétent en réplication malgré la production d’ARNV22,23,24,25,26.
un, Quantification des macrophages et cellules en mousse à reporter mNG (n = 21 images par condition ). b, Quantification des macrophages infectés par le SRAS-CoV-2 ( 24 hpi, n = 20; 48 hpi, n = 24 ) et cellules en mousse ( 24 hpi, n = 20; 48 hpi, n = 18 ). Images représentatives pour un et b afficher les résultats à 48 hpi. Barres d’échelle, 20 μm. L’ANOVA à sens unique avec le test post- hoc de Tukey a été réalisée. Les données sont présentées sous forme de valeurs moyennes ± s.e.m. c, Quantification ARN-FISH de l’ARNv SRAS-CoV-2+ copies cellulaires et fréquence en macrophages (n = 37 ) et cellules en mousse (n = 26). Les données sont présentées sous forme de valeurs moyennes ± s.e.m. Barres d’échelle, 20 um. Mann–Whitney U-test a été effectué. d, Carte thermique des gènes viraux SARS-CoV-2 lit dans les macrophages et les cellules de mousse. e, Carte de chaleur de journal2FC de gènes viraux SARS-CoV-2 dans les macrophages versus cellules en mousse. Le test Wald du paquet DESeq2 a été utilisé pour tester l’importance. Ajusté P les valeurs < 0,05 (FDR = 1%) ont été considérées comme significatives. f, Quantification du titre viral des macrophages infectés par le SRAS-CoV-2 et des surnageants de culture de cellules en mousse (n = 6). Les données sont présentées sous forme de valeurs moyennes ± s.e.m. ANOVA à sens unique suivie par Tukey’s post hoc test a été effectué. g, Diagramme de Venn des DEG dans les macrophages infectés versus non infectés, les cellules en mousse et les gènes partagés. Le test Wald du package DESeq2 a été utilisé pour tester la signification. Les tracés de barres présentent des voies de signalisation régulées classées par leur score combiné. *P < 0,05; **P < 0,01; ***P < 0,001. h, Cartes thermiques du journal2FC des gènes de réponse IFN dans les macrophages et les cellules en mousse. Le test Wald du package DESeq2 a été utilisé pour tester la signification. Réglé P les valeurs < 0,05 ( FDR = 10% ) ont été considérées comme importantes. L’astérisque entre parenthèses indique la comparaison de l’interaction entre l’infection et les termes temporels du modèle. *P < 0,05; **P < 0,01; ***P < 0,001. je, Carte de chaleur de journal2FC de macrophages infectés par le SRAS-CoV-2 versus mousse (n = 3 répétitions biologiques). Le test Wald du paquet DESeq2 a été utilisé pour tester l’importance. Ajusté P les valeurs < 0,05 (FDR = 10%) ont été considérées comme significatives. j, Tracés cinétiques longitudinaux de la réponse combinée de l’IFN et des scores des gènes SARS-CoV-2. Les données sont présentées comme log médian et 25e quartile–75e2FC des cellules infectées par le SRAS-CoV-2 par rapport aux cellules non infectées. Les tests d’hypothèses ont été effectués à l’aide du test de Wilcoxon.
Le SRAS-CoV-2 déclenche des réponses inflammatoires pro-athérogènes
D’après l’observation selon laquelle la réplication du SRAS-CoV-2 a été avortée dans les macrophages et les cellules en mousse et la preuve que la réponse inflammatoire hyperréactive au SRAS-CoV-2 est orchestrée par des macrophages dans d’autres tissus9,14,27, nous avons étudié la réponse immunitaire des macrophages et des cellules en mousse au SRAS-CoV-2. Analyse différentielle de l’expression des gènes des données ARN-seq des macrophages infectés et des cellules en mousse identifiées signatures transcriptionnelles partagées et uniques ( Fig. 2g). Comme prévu, les 1 254 gènes partagés comprenaient les gènes viraux du SRAS-CoV-2. D’autres gènes couramment régulés étaient impliqués dans les réponses antivirales et l’infection au SRAS-CoV-2, y compris les voies de signalisation de l’interféron ( IFN ) et les processus antiviraux par signalisation IFN de type I et II, oligoadénylate synthétase ( OEA ) réponse antivirale, régulation négative de la réplication virale et du cycle de vie viral ainsi que l’activation du complément et la signalisation des cytokines. La signalisation antivirale ISG15, qui amortit la signalisation IFN et a été impliquée dans la réponse hyperinflammatoire associée à la gravité COVID-1927,28, a également été régulé à la hausse dans les deux types de cellules ( Fig. 2g et Fig. Donnée étendue. 3e). Les macrophages infectés ont exprimé une signature transcriptionnelle unique associée à l’activation classique de la cascade du complément, complément la cascade ( Fig. 2g et Fig. Donnée étendue. 3f) ainsi que la signalisation IFN-γ et sa régulation et la signalisation IL-27, qui induisent des gènes dépendants IFN / STAT129 et régulation des voies cytokines ( Fig. 2g). Nous avons également observé une régulation ascendante significative de certains gènes lysosomaux dans les macrophages infectés par rapport aux cellules mousseuses à 2 hpi et 8 hpi ( Données étendues Fig. 3g, h), révélant une réponse lysosomale plus robuste dans les macrophages qui peut contribuer à la clairance virale plus efficace observée dans les macrophages. Notre analyse a montré une augmentation significative des gènes codant pour les protéines impliquées dans l’absorption des lipides dans les macrophages infectés par rapport aux cellules de mousse (Fig. 2g et Extended Data Fig. 3i‘). Ceux-ci inclus CD36, codant pour le récepteur CD36 pour oxLDL, ainsi que LDLR, codant pour le récepteur LDL, qui est la lipoprotéine primaire porteuse de cholestérol dans le plasma et facilite l’absorption des LDL dans les cellules par endocytose. En revanche, les gènes codant des protéines associées à l’efflux lipidique (c’est-à-dire, ABCG1 et ABCA1) ont été significativement régulés à la baisse dans les macrophages infectés par rapport aux cellules de mousse (Données étendues Fig. 3i‘). Les lipides et les gouttelettes lipidiques jouent un rôle crucial dans le cycle de vie viral, y compris le SRAS-CoV-2 (refs. 30,31,32‘). En favorisant l’absorption et l’accumulation des lipides dans les macrophages, le SRAS-CoV-2 peut créer un environnement cellulaire favorable à l’infection virale.
La signature transcriptionnelle unique des cellules en mousse infectée comprenait 570 gènes ( Fig. 2g et Fig. Donnée étendue. 2e). Les voies de signalisation à régulation ascendante supérieure impliquaient la régulation négative du complexe d’histocompatibilité majeur classe I ( MHC-I ), une réponse induite par de nombreux virus pour échapper à la reconnaissance immunitaire33,34. Les cellules en mousse infectées ont également bouleversé les processus et les voies de signalisation impliqués dans la régulation du métabolisme lipidique qui peuvent faciliter l’entrée et la réplication virales32.
L’activation d’une réponse IFN de type I dans les macrophages et les cellules en mousse infectés par le SRAS-CoV-2 était cohérente avec la régulation excessive de plusieurs gènes, avec la réponse la plus forte observée à 8 hpi ( Fig. 2h et Fig. Donnée étendue. 3e). Dans les macrophages, les gènes significativement régulés inclus IRF1, un activateur transcriptionnel de l’IFN-α et de l’IFN-β, ainsi que des gènes induits par l’IFN-α, l’IFN-β et l’IFN-γ; MX1, codant pour une protéine Mx1 liant le GTP qui a une activité antivirale contre les virus à ARN, y compris les références SRAS-CoV-2 (. 35,36); ainsi que STAT1 et STAT2. IRF7, connu pour induire des réponses IFN de type I, et le facteur de restriction virale IFITM1 ( ref. 37), ont également été régulés à la hausse dans les macrophages infectés par le SRAS-CoV-2 ( Fig. 2h). La réponse de l’IFN dans les cellules en mousse infectées par le SRAS-CoV-2 était similaire, bien qu’il y ait eu une déréglementation retardée MX1 qui ne s’est produit qu’à 48 hpi, ce qui correspond à l’accumulation plus élevée d’ARNV et de protéines observée dans les cellules en mousse, et uniquement les cellules en mousse régulées à la hausse IRF4, qui inhibe la signalisation MyD88 et est exprimé en macrophages de type M238. De plus, l’expression de STAT1 et STAT2 n’ont pas été significativement augmentés ( Fig. 2h), suggérant une régulation distincte de la signalisation JAK/STAT induite par l’IFN dans les macrophages infectés. Une comparaison directe entre les macrophages infectés et les cellules de mousse infectées a confirmé une réponse IFN plus forte dans les macrophages par rapport aux cellules de mousse avec une régulation positive significative de JAK1 à 2 hpi et de MAV, codant la protéine de signalisation antivirale mitochondriale essentielle à l’immunité innée antivirale39,40,41, 8 Hpi (Fig. 2i‘).
L’analyse des changements dynamiques de la réponse IFN-I entre les macrophages et les cellules de mousse en utilisant une méthode de notation de signature de gène IFN et SARS-CoV-2 (Méthodes) ont révélé des différences cinétiques importantes dans la réponse à l’IFN et l’expression du gène SARS-CoV-2 entre les macrophages et les cellules de mousse (Fig. 2j). Bien qu’il n’y ait pas de différence significative dans le score du génome du SRAS-CoV-2 entre les macrophages infectés et les cellules en mousse, à 48 hpi, une réduction significative du score du génome du SRAS-CoV-2 a été observée dans les macrophages mais pas dans les cellules en mousse, indiquant une stabilité différentielle de l’ARNV. En parallèle, le score IFN-I a augmenté à la fois dans les macrophages infectés et les cellules en mousse, mais les cellules en mousse ont affiché une baisse significative du score IFN-I à 24 hpi et 48 hpi. Compte tenu des connaissances bien établies que l’IFN de type I réduit l’infection et la réplication du SRAS-CoV-2, favorisant ainsi la clairance virale42,43,44,45, la réponse IFN robuste et soutenue de type I dans les macrophages contribue probablement à une diminution de la persistance virale. De plus, les macrophages et les cellules en mousse ne présentaient pas de différences dans l’activation des voies apoptotiques, telles que la sécrétion de Caspase-8 ( Données étendues Fig. 3j). Plusieurs gènes pro-inflammatoires et pro-athérogènes de cytokine et de chimiokine ont été significativement régulés à la fois dans les macrophages infectés et les cellules en mousse ( Données étendues Fig. 4a, b). Ceux inclus CCL7, TNFSF10 ( également connu sous le nom de TRAIL), CXCL10 et CCL3. Macrophages infectés uniquement régulés CXCL9, CXCL12 et CLEC11A, alors que les cellules de mousse ont été régulées à la hausse TNFA, CCL5 et CCL2.
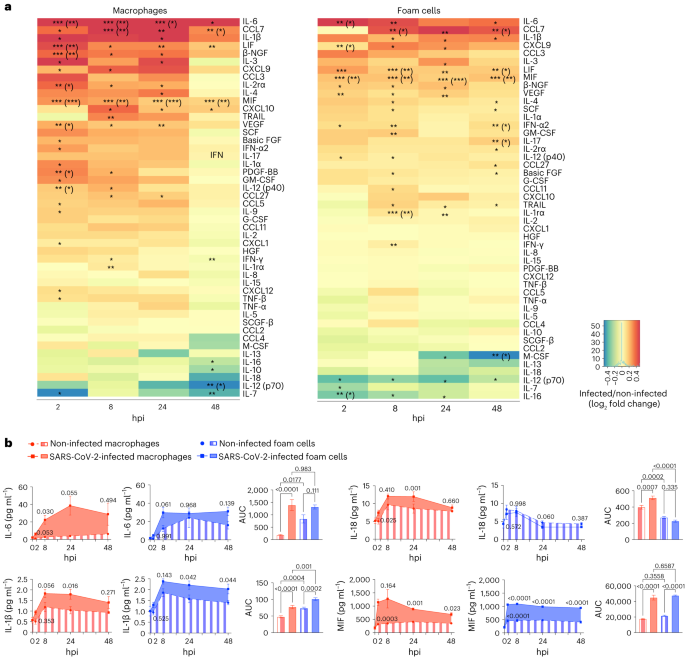
Pour approfondir le profil inflammatoire des macrophages et des cellules en mousse en réponse à l’infection par le SRAS-CoV-2, nous avons quantifié la sécrétion de cytokines et de chimiokines jusqu’à 48 hpi ( Fig. 3 et Fig. Donnée étendue. 4b). Plusieurs cytokines pro-inflammatoires et pro-athérogènes ( par exemple, IL-6, CCL7, IL-1β, β-NGF, IL-3, LIF, MIF, CXCL9, IFN-α et IFN-γ ) ont été libérés par les macrophages infectés et les cellules en mousse ( Fig. 3a). Parmi ceux-ci figurent des cytokines clés connues pour déclencher des événements cardiovasculaires ischémiques, y compris l’IL-6, une cible thérapeutique candidate dans les essais cliniques en cours46, et IL-1β, dont l’inhibition a réduit les événements cardiovasculaires secondaires chez les patients à risque post-infarctus du myocarde élevé47 ( Fig. 3b‘). De plus, la libération du facteur inhibiteur de la migration des macrophages (MIF), une cytokine pro-athérogène et inflammatoire qui a été impliquée dans l’épaississement intima-média, le dépôt lipidique et l’instabilité de la plaque48, a été augmenté dans les deux types de cellules après infection. Cette analyse a également révélé des profils d’expression différentielle des cytokines sécrétées par les macrophages et les cellules de mousse en réponse à l’infection par le SRAS-CoV-2, suggérant des réponses inflammatoires distinctes entre les macrophages et les cellules de mousse. Par example, la cytokine pro-athérogène IL-18 (ref. 49) a été significativement libéré par les macrophages infectés mais pas les cellules de mousse, alors que IFN-2, une cytokine de réponse IFN de type I qui inhibe la réplication virale50, a été significativement libéré par les cellules en mousse infectées mais pas par les macrophages ( Fig. Données étendues. 4b). Nous avons notamment détecté des différences significatives de libération de bon nombre de ces cytokines au fil du temps entre les cellules macrophages et mousse non infectées, suggérant que les différences dans l’état inflammatoire de base de chaque type de cellule influencent leurs réponses au virus ( Fig. 3b et Fig. Donnée étendue. 4b).
un, Carte thermique des cytokines et des chimiokines sécrétées des macrophages et des cellules en mousse infectés par le SRAS-CoV-2. Les données sont affichées sous forme de journal2FC de cellules infectées versus non infectées. P les valeurs ont été calculées par un test t non apparié à deux queues, *P < 0,05; **P < 0,01; ***P < 0,001. Réglé P les valeurs ( Benjamini – La méthode Hochberg ) sont présentées entre parenthèses. b, Les parcelles cinétiques montrent l’ASC des cytokines sécrétées par les macrophages et cellules en mousse infectés par le SRAS-CoV-2 par rapport aux cellules non infectées (n = 4 répétitions biologiques, doublons techniques ). Pour les comparaisons AUC, l’ANOVA unidirectionnelle après le test de comparaisons multiples de Tukey a été effectuée. Les différences cinétiques ont été évaluées par l’ANOVA bidirectionnelle suivie du test de comparaisons multiples de Sidak. Les données sont présentées comme moyennes ± s.e.m. Les placettes de bar montrent la quantification de l’ASC pour chaque cytokine. Analyse statistique ANOVA unidirectionnelle après le test de comparaisons multiples de Tukey.
Le SRAS-CoV-2 stimule l’inflammation des explants vasculaires athérosclérotiques
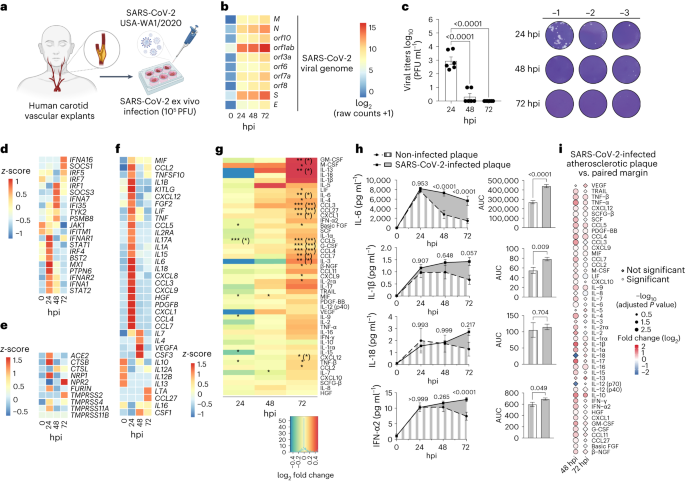
Déterminer si la réponse pro-inflammatoire des cellules macrophages et moussants au SRAS-CoV-2 que nous avons observée in vitro se produit également dans les tissus vasculaires humains, nous avons infecté des explants vasculaires athérosclérotiques humains avec l’isolat SRAS-CoV-2 USA WA1 // 2020 ( Fig. 4a). ARNv SRAS-CoV-2 codant pour la pointe des protéines structurelles ( S ), enveloppe ( E ), membrane ( M ) et nucléocapside ( N ), ainsi que des cadres de lecture ouverts ( ORF ) codant pour les protéines accessoires non structurelles, ont été détectables dans les plaques infectées dès 24 hpi et jusqu’à 72 hpi ( Fig. 4b). L’expression des protéines S et N et les particules de type virus ont été observées dans des plaques athérosclérotiques infectées ( Fig. Données étendues. 5a, b). Cependant, le titre viral a diminué au fil du temps sans virus infectieux isolé des milieux conditionnés des cellules ou des tissus infectés à l’aide d’un test de plaque jusqu’à 72 hpi, suggérant une réplication avortée dans les explants vasculaires ( Fig. 4c). Quoi qu’il en soit, l’infection au SRAS-CoV-2 a induit une forte réponse transcriptionnelle IFN de type I dans les plaques infectées reflétée par la régulation précoce des facteurs de transcription et des gènes impliqués dans la réponse aux infections virales, telles que IRF7, JAK1 et IFITM1 à 2 hpi; IFNAR, IRF4, IRF1, MX1, PTPN6, IFNA1, STAT1 et STAT2 à 24 hpi; IRF1, IFNA7 et IFI35 à 48 hpi; et IFNA16 à 72 hpi. Généralités impliquées dans la régulation négative de la signalisation IFN ( c’est-à-dire, SOCS1 et SOCS3) a également été régulé à la hausse, reflétant probablement l’activation de la signalisation réglementaire ( Fig. 4d). Fait intéressant, l’infection au SRAS-CoV-2 a déclenché l’expression de récepteurs viraux et de facteurs d’entrée, tels que ACE2, PNR1, FURIN, TMPRSS4, TMPRSS11A et CTSB à 24 hpi, ce qui suggère que le virus facilite sa propre entrée dans les cellules hôtes ( Fig. 4e). Le SRAS-CoV-2 a également lancé une réponse pro-inflammatoire transcriptionnelle qui a largement récapitulé celle observée dans les macrophages et les cellules en mousse cultivés. Cela comprenait la régulation des cytokines pro-athérogènes, comme IL1B, IL6, MIF, ILB, TNF, IL7 et CCL5, ainsi que des chimiokines, telles que CCL2, CCL3, CCL4, CCL5, CXCL9, CCL27, CCL7, CCL6, CXCL1, CXCL8, CXCL9 et CXCL12. Cytokines anti-inflammatoires, telles que IL10 et IL13, ont été déréglementés, soutenant davantage une forte réponse inflammatoire pro-athérogène à l’infection par le SRAS-CoV-2 dans les plaques athérosclérotiques humaines ( Fig. 4f et Fig. Donnée étendue. 5c). L’analyse du sécrétome des plaques infectées a révélé des changements similaires de protéines pro-inflammatoires ( Fig. 4g). Les plaques infectées par le SRAS-CoV-2 ont libéré plusieurs cytokines et chimiokines pro-athérogènes, dont IL-6, IL-1β et IFN-α2, ainsi que CCL2, CCL3, CCL4 et CCL7 ( Fig. 4g‘). La libération de cytokines et de chimiokines était sensiblement plus élevée à 72 hpi. Cependant, seuls l’IL-6, l’IL-1, l’IFN-levante2 et le CCL3 ont été sécrétés à des quantités significativement plus élevées au fil du temps calculées comme surface sous la courbe (ASC) (Fig. 4h et Extended Data Fig. 5d‘). Une réponse inflammatoire plus forte a été observée dans les plaques athérosclérotiques infectées par le SRAS-CoV-2 par rapport aux marges chirurgicales appariées non athérosclérotiques, comme le montre la libération significativement plus élevée de plusieurs cytokines (par exemple, IFN-similaire, IFN-2, IL-1, IL-17, TNF-17, TNF-F, CCL3, CCL4 et CCL7) à partir de plaques infectées principalement à 48 hpi (Fig. 4i). Ces résultats suggèrent que l’infection au SRAS-CoV-2 déclenche une réponse immunitaire hyperactivée principalement dans les lésions athérosclérotiques, une réponse qui pourrait contribuer au risque accru d’événements cardiovasculaires ischémiques chez les patients atteints de COVID-19 atteints d’athérosclérose sous-jacente.
a, Schémas de l’approche expérimentale de l’infection d’explants vasculaires carotides humains avec le SRAS-CoV-2. b, 2, Carte thermique des lectures virales du SRAS-CoV-2 dans les explants vasculaires carotides à l’inclusion (0 hpi), 24 hpi, 48 hpi et 72 hpi. c, Quantification du titre viral infectieux des surnageants de culture de plaque carotidienne infectés par le SRAS-CoV-2 (n = 3 échantillons biologiques, duplicatas techniques). Les données sont présentées sous forme de valeurs moyennes ± s.e.m. ANOVA à sens unique suivie par Tukey’s post hoc test a été effectué. d, Carte de chaleur montrant le standardisé zexpression marquée des gènes de réponse à l’IFN dans des échantillons vasculaires carotides infectés par le SRAS-CoV-2 à différents moments après l’infection. e, Carte de chaleur de standardisé z-expression marquée de récepteurs viraux hôtes sélectionnés et de facteurs d’entrée dans des échantillons vasculaires carotides humains infectés par le SRAS-CoV-2. f, Carte thermique de normalisé z-expression génique marquée des gènes de cytokine et de chimiokine dans les explants vasculaires carotides humains infectés par le SRAS-CoV-2 à différents moments après l’infection. g, Carte thermique des cytokines et des chimiokines sécrétées à partir de plaques athérosclérotiques humaines infectées par le SRAS-CoV-2. Les données sont affichées sous forme de journal2FC d’échantillons infectés versus non infectés. P les valeurs ont été calculées par des paires à deux queues t-test, *P < 0,05; **P < 0,01; ***P < 0,001. P les valeurs entre parenthèses ont été ajustées à l’aide de la méthode Benjamini – Hochberg. h, Les parcelles cinétiques montrent l’ASC des cytokines et des chimiokines sécrétées par des plaques carotides non infectées ou infectées par le SRAS-CoV-2 (n = 3 donneurs, doublons techniques ). Les données sont présentées sous forme de valeurs moyennes ± s.e.m. Analyse statistique ANOVA bidirectionnelle après le test de comparaisons multiples de Sidak. En couple t-test a été effectué pour comparer l’ASC de deux groupes. je, Tracé montrant l’expression relative de cytokines sécrétées et de chimiokines entre la plaque athérosclérotique infectée par le SRAS-CoV-2 par rapport aux marges vasculaires. L’expression relative est représentée dans le journal2Échelle colorée FC. La signification statistique est exprimée en taille de point. Les valeurs statistiquement significatives sont représentées en cercles et les changements non significatifs sont représentés en diamants. P les valeurs ont été calculées par un apparié à deux queues t-test. P les valeurs ont été ajustées à l’aide de la méthode Benjamini – Hochberg.
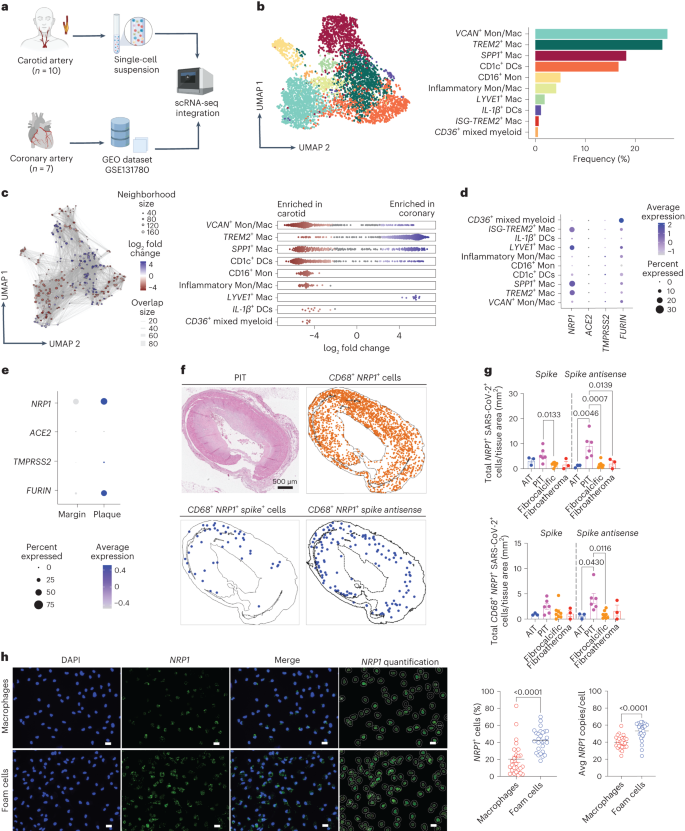
PNR1+ les macrophages augmentent la sensibilité de la plaque au SRAS-CoV-2
Pour élucider la susceptibilité vasculaire à l’infection par le SRAS-CoV-2, nous avons évalué l’expression des principaux récepteurs d’entrée virale et des cofacteurs dans l’aorte, artères coronaires et tibiales à l’aide de données d’expression génique accessibles au public dans le cadre du projet Genotype-Tissue Expression (GTEx) (https://gtexportal.org/home/‘). Le poumon, le tissu cardiaque et le sang total ont également été inclus dans cette analyse (Données étendues Fig. 6a, b‘). Nous nous sommes concentrés spécifiquement sur ACE2, encodant le premier récepteur rapporté pour l’entrée du SRAS-CoV-2 dans les cellules humaines; neuropilines (PNR1 et PNR2); et les protéases TMPRSS2, FURINE, cathepsine B (CTSB) et cathepsine L (CTSL), nécessaire pour cliver la protéine S pour l’entrée et la réplication virales51,52,53,54. L’analyse du séquençage de l’ARN en vrac ( RNA-seq ) a montré un modèle d’expression similaire pour ACE2, PNR1, PNR2, FURIN et CTSB et CTSL dans l’aorte, les artères coronaires et tibiales par rapport au poumon, à l’exception de TMPRSS2, qui a été exprimé à des niveaux inférieurs dans les artères ( Données étendues Fig. 6b).
Étudier l’expression cellulaire du récepteur du SRAS-CoV-2 et les facteurs d’entrée dans le tissu athérosclérotique humain, nous avons effectué une analyse scRNA-seq intégrée des plaques de carotide humaine de 10 patients subissant une endartériectomie carotidienne (AEC) et des données coronariennes athérosclérotiques obtenues à partir de sept échantillons coronaires de quatre cas de transplantation cardiaque accessibles au public dans le GEO (GSE131780) (Fig. 5a‘)15. Les deux ensembles de données ont été fusionnés en utilisant l’algorithme Harmony, résultant en 16 sous-groupes de cellules immunitaires qui correspondaient à toutes les populations immunitaires majeures infiltrant les plaques athérosclérotiques humaines (Données étendues Fig. 6c‘). Les récepteurs d’entrée du SRAS-CoV-2 et les facteurs d’entrée de l’hôte tels que PNR1, 2, un récepteur du SRAS-CoV-2 qui peut lier la protéine S clivée par FURIN pour faciliter l’entrée virale du SRAS-CoV-2, et les protéases CTSB et CTSL étaient fortement exprimés dans les sous-groupes myéloïdes, alors que ACE2 et les protéases à sérine transmembranaires TMPRSS2, TMPRSS4, TMPRSS11A et TMPRSS11B étaient soit indétectables, soit exprimés à de faibles niveaux (Données étendues Fig. 6d‘). Sur la base de cette observation, nous avons sous-clusté les cellules myéloïdes (Fig. 5b et Extended Data Fig. 6e) et identifié deux groupes de cellules dendritiques ( DC ), trois groupes de monocytes / macrophages, un groupe de cellules myéloïdes mixtes et quatre groupes de macrophages/cellules en mousse annotées sur la base de l’expression de marqueurs canoniques ( Données étendues Fig. 6f). Pour identifier les différences significatives dans l’abondance des cellules myéloïdes entre les artères carotides et coronaires, nous avons effectué des tests d’abondance du quartier différentiel de Milo55. Cette analyse a révélé que TREM2+ les macrophages ont été enrichis en tissu coronaire, tandis que VCAN+ monocytes / macrophages et CD1c+ Les CC ont été enrichis en échantillons de carotides. CD16+ monocytes, monocytes / macrophages inflammatoires, IL1B+ DC et CD36+ les cellules myéloïdes mixtes étaient exclusivement présentes dans les échantillons de carotides, alors que LYVE1+ des macrophages étaient présents dans les coronaires. SPP1+ des macrophages étaient présents dans les deux tissus ( Fig. 5c). Dans l’ensemble, PNR1 a été fortement exprimé en TREM2+, SPP1+, LYVE1+ et gène stimulé par l’IFN (ISG)+TREM2 + macrophages, grappes qui ont également exprimé FURIN. ACE2 et TMPRSS2 étaient indétectables dans les cellules myéloïdes analysées (Fig. 5d‘). PNR1 et FURINE l’expression génique était plus élevée dans les plaques athérosclérotiques humaines que dans les marges normales appariées (Fig. 5e), suggérant un rôle clé pour le PNR-1 dans la médiation de l’infection par le SRAS-CoV-2 du système vasculaire athérosclérotique. L’analyse spatiale RNA-FISH de spécimens d’autopsie coronarienne humaine de patients atteints de COVID-19 a confirmé que les macrophages s’expriment PNR1 (CD68+PNR1+ les cellules) ont infiltré les lésions coronaires et que ces cellules ont également exprimé le vRNA S du SRAS-CoV-2 et le brin antisens du gène S, indiquant une réplication virale (Fig. 5f, g‘). Un nombre plus élevé de PNR1+ des macrophages exprimant le brin antisens du gène S ont été trouvés dans les lésions coronaires PIT, ce qui correspond à la plus grande susceptibilité des lésions PIT à l’infection (Fig. 5g‘).
a, scRNA-seq de la carotide humaine (n = 10) et coronaire (GSE131780() (n = 7) échantillons de tissus. b, visualisation UMAP des sous-groupes de cellules myéloïdes à partir d’échantillons coronariens (1 960 cellules) et carotidiens (2 900 cellules). Le diagramme à barres montre la fréquence de chaque groupe myéloïde. c, Graphique de voisinage des résultats du test d’abondance différentielle MiloR. Les nœuds représentent les quartiers, colorés par leur journal2FC entre les échantillons de carotide (rouge) et coronaire (bleu). Les quartiers d’abondance non différentiels sont en blanc (FDR = 10%), et la taille des nœuds reflète le nombre total de cellules dans chaque quartier. Les parcelles de Beeswarm montrent le journal2distribution FC des voisinages entre le type de tissu (FDR = 10%). d, Dot plot de l’expression du gène moyen du facteur d’entrée viral SARS-CoV-2 et pourcentage d’expression dans chaque sous-groupe myéloïde. e, des parcelles ponctuelles montrant la fréquence des cellules exprimant les facteurs d’entrée virale du SRAS-CoV-2 colorés par l’expression moyenne dans les lésions de la plaque athéroscléreuse et les marges de vascularisation appariées. f, Images représentatives de H&E et analyse spatiale de l’échantillon coronaire PIT montrant l’emplacement de CD68+PNR1+ cellules, CD68+PNR1+ SARS-CoV-2 S+ ou S antisens+ cellules. g, Parcelles de barres montrant le total PNR1+ ARNv du SRAS-CoV-2+ et CD68+PNR1+ ARNv du SRAS-CoV-2+ cellules normalisées par zone tissulaire (mm2) dans AIT (n = 3), FOSSE (n = 6), fibrocalciques (n = 8) et fibroathérome (n = 3) coronaires. h, Images représentatives et quantification RNA-FISH de la fréquence de PNR1+ cellules et moyenne PNR1 copies par cellule dans les macrophages non infectés (n = 27) et cellules de mousse (n = 28). Barres d’échelle, 20 um. L’analyse statistique a été réalisée en utilisant des Student’s non appariés à deux queues t-test. Avg, moyenne; Mac, macrophages; Mon, monocytes.
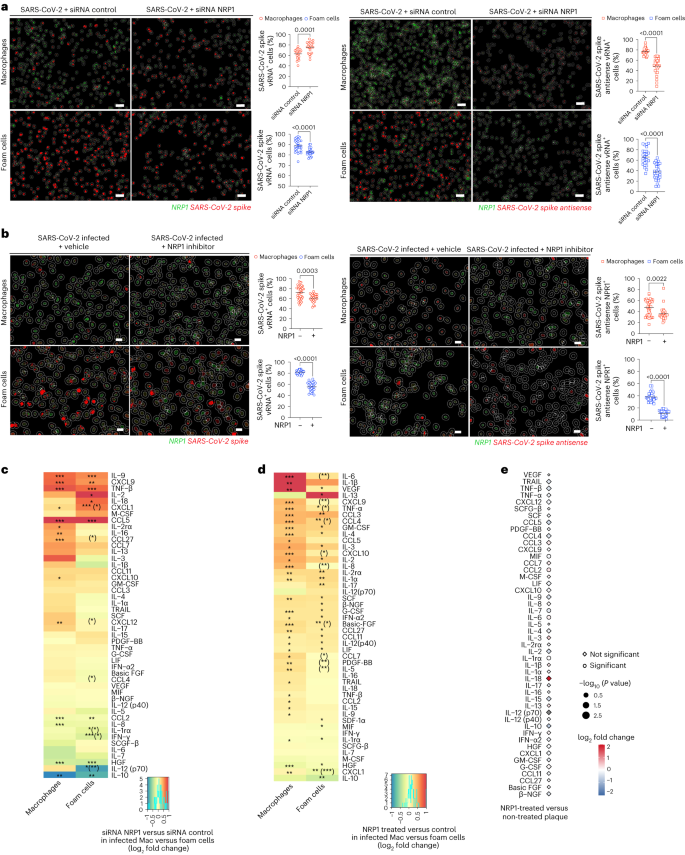
Compte tenu de cela PNR1 a été fortement exprimé en TREM2+ macrophages, qui sont considérés comme des macrophages de plaque mousseux56, et il était significativement plus élevé dans les cellules de mousse par rapport aux macrophages in vitro (Fig. 5h), nous avons effectué des expériences supplémentaires en utilisant l’ARN silencieux (siRNA) pour inhiber spécifiquement l’expression de NRP-1 et étudier son impact sur l’infection par le SRAS-CoV-2 (Fig. 6a, b et Extended Data Fig. 7a–f‘). Nous avons réalisé la réduction efficace (~80%) de PNR1 expression génique dans les macrophages et les cellules de mousse, confirmée par western blotting et analyse de l’ARNscope (Données étendues Fig. 7a–d et Complémentaire Fig. 2‘). Sur PNR1 en réduisant au silence les cellules infectées, nous avons observé une diminution significative de la fréquence de l’antisens S du SRAS-CoV-2+ macrophages et cellules de mousse ainsi qu’une fréquence réduite de SARS-CoV-2 S+ cellules de mousse (Fig. 6a et Extended Data Fig. 7e‘). Ces résultats ont été confirmés en utilisant EG00229, une petite molécule qui empêche la liaison du SRAS-CoV-2 au domaine B1 du PNR-1 et réduit l’infection des lignées cellulaires par le SRAS-CoV-253,54 (Fig. 6b et Extended Data Fig. 7f‘).
un, Images représentatives de la quantification ARN-FISH basée sur l’IA montrant PNR1 ARN, SRAS-CoV-2 S et S antisens+ cellules. Barres d’échelle, 50 μm. Quantification de la fréquence du SRAS-CoV-2 S+ et SRAS-CoV-2 S antisense+ cellules en macrophages et cellules en mousse traitées avec un contrôle de l’ARNsi non ciblant ou siRNA NRP1 à 24 hpi. Les données sont présentées sous forme de valeurs moyennes ± s.e.m. Une analyse statistique a été réalisée à l’aide d’un étudiant à deux queues non jumelé t-test. b, Images représentatives de la quantification ARN-FISH montrant PNR1 ARN, SRAS-CoV-2 S et S antisens+ cellules. Barres d’échelle, 20 μm. Quantification de la fréquence du SRAS-CoV-2 S+ et antisens SARS-CoV-2 S+ cellules dans les macrophages et les cellules de mousse avec et sans inhibition de NRP1 (EG00229 trifluoroacétate) à 24 hpi. c, Carte thermique des niveaux de cytokines et de chimiokines sécrétées de manière différentielle à partir de macrophages infectés par le SRAS-CoV-2 et de cellules de mousse (n = 4–5) après traitement avec contrôle siRNA ou siRNA NRP1 à 24 hpi. Ajusté P les valeurs < 0,05 ont été considérées comme significatives. Asterisk indique un ajustement P valeur < 0,05 pour la comparaison du SRAS-CoV-2 infecté et traité avec le siRNA NRP1 contre le contrôle infecté et le siRNA. Les astérisques entre parenthèses indiquent une valeur nominale P < 0,05 pour la comparaison entre les macrophages et les cellules en mousse, *P < 0,05; <TAG1>; **P < 0,01; <TAG1>; ***P < 0,001. d, Carte thermique des niveaux de cytokines et de chimiokines sécrétées de manière différentielle à partir de macrophages infectés par le SRAS-CoV-2 et de cellules de mousse après blocage du PNR1 (EG00229 trifluoroacétate). Les résultats sont affichés sous forme de journal2FC entre les conditions infectées et non traitées. Ajusté P les valeurs < 0,05 ont été considérées comme significatives. Asterisk indique un ajustement P valeur < 0,05 pour la comparaison des conditions infectées et traitées par le SRAS-CoV-2 par rapport aux conditions infectées et traitées par le véhicule. Les astérisques entre parenthèses indiquent une valeur nominale P valeur < 0,05 pour la comparaison entre macrophages et cellules en mousse, *P < 0,05; <TAG1>; **P < 0,01; <TAG1>; ***P < 0,001. e, Tracé montrant l’expression relative de cytokines et de chimiokines sécrétées provenant du blocage du PNR1 ( EG00229 trifluoroacétate ) traités versus plaques athérosclérotiques infectées par le SRAS-CoV-2 non traitées à 48 hpi. L’expression relative est représentée dans le journal2Échelle colorée FC. Les cercles représentent des résultats statistiquement significatifs et les changements non significatifs sont représentés comme des diamants.
Nous avons ensuite demandé si PNR1 le silence ou le blocage à l’aide d’EG00229, qui réduisait l’infection des macrophages et des cellules en mousse in vitro, réduirait la réponse inflammatoire au SRAS-CoV-2. Les résultats ont révélé que le silencieux NRP1 augmentait l’état inflammatoire de base des macrophages non infectés et, dans une moindre mesure, des cellules en mousse, comme en témoigne la libération de plusieurs cytokines et chimiokines ( Données étendues Fig. 8a). Notamment, cet état inflammatoire a persisté sous l’infection par le SRAS-CoV-2, infecté PNR1-les cellules silencieuses présentant des niveaux plus élevés de cytokines induites par le virus, telles que l’IL-6 et le MIF, par rapport aux cellules non infectées du PNR1 ( Fig. 6c et Fig. Donnée étendue. 8a). Dans les cellules en mousse, le silencieux NRP-1 a eu des effets plus modestes sur les cellules non infectées et infectées, la libération de cytokines inflammatoires étant principalement due à l’infection par le SRAS-CoV-2 ( Fig. 6 et Fig. Donnée étendue. 8a). Les résultats sont cohérents avec les fonctions anti-inflammatoires du PNR-1 dans les cellules myéloïdes et l’augmentation de l’inflammation lorsque l’expression du PNR1 est perdue57,58. Fait intéressant, l’utilisation d’EG00229 a donné des schémas inflammatoires similaires, mais certaines différences peuvent suggérer des effets hors cible potentiels associés à cet inhibiteur ( Fig. 6d). Étudier la modulation potentielle du TGF-β par PNR1 l’inhibition, nous avons examiné la sécrétion de TGF-E dans les macrophages infectés et les cellules de mousse (Extended Data Fig. 8b‘). Bien que, dans les macrophages, l’expression de TGF-sondage n’ait pas été affectée par l’infection par le SRAS-CoV-2, et l’inhibition de NRP-1 n’a pas influencé son expression, nous avons observé une augmentation des niveaux de TGF-stimulant dans les cellules de mousse infectées en raison de l’infection par le SRAS-CoV-2 qui a été partiellement réduite par l’inhibition du PNR-1. Le blocage du PNR-1 a augmenté la libération d’IL-6 et de CCL2 sécrétées par le tissu d’explant vasculaire athérosclérotique humain infecté par le SRAS-CoV-2. La libération d’IL-1 et d’IL-18 a également été augmentée mais pas de manière significative (Fig. 6e). Ensemble, ces résultats suggèrent que les plaques athérosclérotiques sont particulièrement sensibles à l’infection par le SRAS-CoV-2, qui déclenche une réponse immunitaire hyperactivée qui pourrait contribuer au risque accru d’événements cardiovasculaires ischémiques chez les patients atteints de COVID-19 avec athérosclérose sous-jacente. Bien que l’inhibition du facteur d’entrée de l’hôte NRP-1 exprimée par des macrophages infiltrant des lésions coronariennes chez l’homme ait réduit l’infection au SRAS-CoV-2, elle a également induit une forte réponse inflammatoire, indiquant que des stratégies alternatives seront nécessaires pour prévenir l’infection par le SRAS-CoV-2 des vaisseaux coronariens et ses conséquences en aval.
Discussion
Bien que le SRAS-CoV-2 soit considéré comme un virus respiratoire, les patients atteints de COVID-19 se sont révélés présenter un risque accru de complications cardiovasculaires, y compris un infarctus du myocarde et un accident vasculaire cérébral. Notre étude fournit des preuves de la présence du SRAS-CoV-2 dans le système vasculaire coronarien humain et démontre un tropisme viral pour les macrophages des lésions vasculaires chez les personnes atteintes de COVID-19 sévère. Nous avons trouvé des preuves de réplication du SRAS-CoV-2 dans tous les coronaires d’autopsie humaine analysés, quelle que soit leur classification pathologique, bien que la réplication virale ait été la plus élevée dans les lésions coronariennes PIT — les lésions précoces qui progressent vers des plaques athérosclérotiques plus avancées11,12. Il est bien établi que, à mesure que les lésions progressent vers des stades plus avancés, tels que les fibroathéromes et les plaques fibrocalciques, les macrophages et les cellules moussantes deviennent dysfonctionnels en raison d’une exposition prolongée aux lipides et d’un microenvironnement inflammatoire11,12. Dans les lésions PIT précoces, les macrophages qui tentent d’éliminer les lipides inflammatoires (par exemple, les oxLDL) accumulent un excès de cholestérol et de triglycérides dans les gouttelettes lipidiques cytoplasmiques12 peut être plus sujet aux infections virales. Le SRAS-CoV-2 a montré un tropisme plus fort pour les lésions artérielles que la graisse périvasculaire correspondante, qui était liée au degré d’infiltration des macrophages, compatible avec la réplication virale plus élevée dans les lésions PIT et les fibroathéromes, où les macrophages sont plus répandus. D’autres ont déjà signalé la présence d’ARNv du SRAS-CoV-2 dans le cœur et l’aorte ainsi que dans d’autres organes distants8,14,59,60. Nos données démontrent de manière concluante que le SRAS-CoV-2 est capable d’infecter et de reproduire dans les macrophages dans le vasculature coronaire des patients atteints de COVID-19. De plus, le SRAS-CoV-2 se reproduit préférentiellement dans les cellules en mousse par rapport à d’autres macrophages, en raison d’une réponse IFN réduite de type I qui conduit à l’ARNV et à l’accumulation de protéines virales, suggérant que ces cellules pourraient servir de réservoir de débris viraux du SRAS-CoV-2 dans la plaque athérosclérotique. Nos résultats démontrent que, bien que la réponse IFN de type I soit restée élevée dans les macrophages infectés par le SRAS-CoV-2, indiquant un état antiviral soutenu, les cellules en mousse infectées ne présentaient qu’une expression transitoire de l’expression du gène IFN de type I. Associé à l’expression réduite de gènes lysosomaux et à une réponse inflammatoire plus faible à l’infection par le SRAS-CoV-2,la réduction de l’IFN de type I peut contribuer à la persistance du SRAS-CoV-2 observée dans les spécimens d’autopsie athéroscléreuse analysés. Les macrophages résidant dans le tissu vasculaire peuvent subir l’auto-renouvellement, contribuant à leur entretien à long terme61, et, dans les études de transplantation cardiaque, il a été démontré que les macrophages cardiaques ont une demi-vie de 8,8 ans62. Par conséquent, il est possible que les macrophages agissent comme des réservoirs d’ARNv SRAS-CoV-2 dans des plaques athérosclérotiques. Nous avons également identifié des VSMC infectés dans des échantillons d’autopsie coronaire. Bien que le nombre de VSMC infectés soit inférieur à celui des macrophages infectés, les VSMC ont une demi-vie de 270 – 400 d63 et peut contribuer à la persistance du SRAS-CoV-2 dans la paroi artérielle.
L’infection à SRAS-CoV-2 des macrophages et des cellules en mousse a favorisé une forte réponse inflammatoire caractérisée par la libération de cytokines impliquées à la fois dans la pathogenèse de l’athérosclérose et le risque accru d’événements cardiovasculaires, tels que l’accident vasculaire cérébral et l’infarctus du myocarde46,47,64. En utilisant un modèle ex vivo d’infection virale d’explants vasculaires humains, nous avons constaté que le tissu athérosclérotique pouvait être directement infecté par le SRAS-CoV-2, confirmation de notre observation dans les tissus des patients atteints de COVID-19. Comme dans les macrophages cultivés et les cellules de mousse, l’infection par le SRAS-CoV-2 du tissu vasculaire a déclenché une réponse inflammatoire et induit la sécrétion de cytokines pro-athérogènes clés, telles que l’IL-6 et l’IL-1. Considérant que l’inflammation favorise la progression de la maladie et contribue à la rupture, nos résultats fournissent une base moléculaire sur la façon dont l’infection par le SRAS-CoV-2 des lésions coronariennes peut contribuer aux manifestations cardiovasculaires aiguës du COVID-19, telles que l’infarctus du myocarde3,5. L’infection par le SRAS-CoV-2 des coronaires n’était pas liée aux caractéristiques cliniques préexistantes, au stade de COVID-19 selon les jours de maladie, à la durée de l’hospitalisation au moment du décès ou aux comorbidités. Cependant, nous avons trouvé une accumulation plus élevée d’ARNv antisens S et S du SRAS-CoV-2 dans le système vasculaire coronaire des trois patients présentant des manifestations cardiovasculaires ischémiques aiguës, y compris un infarctus du myocarde postérieur (Pt. 1) et infarctus du myocarde de type II (Pt. 2 et Pt. 3). Bien qu’aucune preuve d’occlusion coronarienne n’ait été détectée à l’autopsie chez deux patients ayant reçu un diagnostic clinique d’infarctus du myocarde, ces données suggèrent que l’infection coronarienne SARS-CoV-2 pourrait augmenter le risque cardiovasculaire. Une analyse systématique de GTEx, un ensemble de données d’expression génique multi-tissus qui comprend des donneurs décédés d’une maladie cérébrovasculaire (>22% ) et les maladies cardiaques ( > 40% ), ont révélé un schéma d’expression similaire des récepteurs et cofacteurs du SRAS-CoV-2 dans le système vasculaire humain comme celui trouvé dans les poumons. En particulier, bien que ACE2 l’expression était faible dans l’aorte et l’artère tibiale, ses niveaux d’expression dans l’artère coronaire sont similaires à ceux du poumon, suggérant que le système vasculaire coronaire pourrait être plus sensible à l’infection virale par le SRAS-CoV-2 que les autres lits vasculaires. Au niveau unicellulaire, l’expression des récepteurs et des facteurs du SRAS-CoV-2 a confirmé nos résultats d’autopsie du système vasculaire coronarien. Bien que ACE2 l’expression n’était pas détectable par scRNA-seq, PNR1 et FURINE ils ont été fortement exprimés en deux TREM2+ les amas de macrophages, connus pour correspondre aux macrophages mousseux de la plaque56, ainsi que dans les groupes de SSP1+ macrophages et LYVE1+ macrophages. Nous avons également constaté que le SRAS-CoV-2 infecte le PNR1+ macrophages dans le tissu coronaire humain à l’autopsie et que la réplication virale était plus grande dans NRP1+ macrophages présents dans les lésions PIT. Expérimentalement, les deux font taire PNR1 gène et inhibiteur spécifique de l’interaction entre le domaine b1 de NRP-1 et le CendR SARS-CoV-2 S153,54 l’infectiosité réduite du SRAS-CoV-2 des macrophages primaires humains et des cellules en mousse, confirmant que l’infection par le SRAS-CoV-2 des cellules macrophages et en mousse dépend en partie du PNR-1. Cependant, une réponse aberrante pro-inflammatoire associée à l’inhibition du PNR-1 limite l’utilisation thérapeutique potentielle de l’inhibiteur du PNR-1. Cet effet pro-inflammatoire, bien qu’inattendu dans le contexte d’une infection au SRAS-CoV-2, est conforme aux constatations antérieures d’un rôle protecteur du PNR-1 dans la septicémie et de la libération accrue de cytokines pro-inflammatoires ( par exemple, IL-6 ) des macrophages nuls NRP-158.
Dans l’ensemble, nos données démontrent que le SRAS-CoV-2 se reproduit dans les macrophages au sein des coronaires humains des patients décédés d’un COVID-19 sévère. Notre étude se limite à l’analyse d’une petite cohorte de personnes âgées atteintes de COVID-19 et d’athérosclérose préexistante et d’autres conditions médicales et comorbidités. Par conséquent, nos observations ne peuvent pas être extrapolées à des individus plus jeunes et en bonne santé. Notre étude est également limitée aux cas survenus au cours des premières phases de la pandémie de COVID-19, et les résultats que le SRAS-CoV-2 reproduit dans le vasculature coronarien athérosclérotique ne concernent que les souches virales qui ont circulé à New York entre mai 2020 et mai 2021. Malgré ces limites,notre étude met en évidence la réponse hyperinflammatoire orchestrée par les macrophages de plaque infectés par le SRAS-CoV-2 et les cellules de mousse comme un lien mécaniste entre l’infection des vaisseaux coronaires athérosclérotiques et les complications cardiovasculaires aiguës du COVID-19.
Méthodes
L’approbation éthique pour l’utilisation de spécimens d’autopsie humaine a été obtenue auprès du conseil d’examen institutionnel (IRB) de NYU Langone Health (NYULH) (IRB i21-01587) conformément à toutes les réglementations éthiques pertinentes. Les patients soumis au CEA qui ont signé un consentement éclairé écrit ont été inscrits à l’étude ATHERO-IN approuvée par les IRB de l’École de médecine d’Icahn au Mont Sinaï (IRB 11–01427) et NYULH (IRB i21-00429). Les informations démographiques et les antécédents cliniques rapportés sont dépersonnalisés et couverts par le consentement des patients’ à publier de telles données conformément aux normes IRB 11–01427 et IRB i21-00429.
Échantillons d’autopsie coronaire de patients atteints de COVID-19
Spécimens d’artère coronaire fixés à la formol et en paraffine (n = 27 ) de huit patients décédés diagnostiqués avec COVID-19 ont été obtenus de NYULH et du NYU Grossman School of Medicine’s Center for Biospecimen Research and Development ( CBRD ). Des informations démographiques et des antécédents cliniques ont été obtenus à partir des dossiers médicaux de l’hôpital ( Tableau supplémentaire 1). Des données sur l’évolution clinique de l’infection et la pathologie associée au COVID-19 ont été obtenues à la fois dans les dossiers médicaux de l’hôpital et dans les rapports de pathologie d’autopsie ( Tableau supplémentaire 2).
NAccope hybridation in situ
Le test RNAscope 4-plex a été effectué sur des sections de tissu coronaire à l’aide du kit de réactif multiplex LS et du kit d’ancrage LS 4-plex RNAG ( ACD Bio-Techne ). Des sondes d’ARNscope spécifiques au SRAS-CoV-2 ont été utilisées pour visualiser l’ARNv du SRAS-CoV-2 codant pour la protéine S et pour détecter une réplication virale directe à l’aide d’une sonde sensorielle qui cible le brin antisens de le gène S. Des sondes de contrôle négatives et positives ont été utilisées pour évaluer l’intégrité de l’ARN tissulaire. Sondes d’ARNscope utilisées pour détecter CD68, ACTA2 et PNR1 les transcriptions, le gène viral SRAS-CoV-2 S, le brin antisens du gène S et d’autres réactifs sont détaillés dans le tableau supplémentaire 5. Les sections de tissus ont été numérisées à un grossissement de ×40 dans le Vectra Polaris Automated Quantitative Pathology Imaging System à l’aide d’un flux de travail MOTIF. Pour les expériences in vitro, macrophages dérivés de cellules mononucléées du sang périphérique humain, cellules de mousse dérivées de macrophages, macrophages, les VSMC aortiques primaires et les VSMC chargées en cholestérol ont été infectées par le SRAS-CoV-2 avant d’effectuer le protocole RNAscope ISH Multiplex Fluorescent V2 Assay selon les instructions du fabricant’s. Les images ont été acquises avec un microscope Keyence BZ-X800.
Analyse d’hybridation in situ de RNAscope
Les images ont été annotées dans Phenochart visionneuse de diapositives entières (Akoya Biosciences). Les fichiers ‘.qptiff’ ont été estampillés pour le lot InForm sur Phenochart (version 1.1). Le démixage spectral de toutes les images a été effectué à l’aide de l’algorithme automatisé InForm’s. Les images estampillées ont été traitées par InForm (PerkinElmer, version 2.6), et les fichiers ‘component_data.tif’ ont été exportés pour la quantification en utilisant la plate-forme d’analyse d’image HALO (version 3.5.3577) et HALO AI (version 3.6.4134) (Indica Labs) en utilisant le module classificateur, module classificateur de réseau neuronal et module d’analyse spatiale. Les algorithmes utilisés étaient Area Quantification (version 2.4.2 et version 2.4.3), Area Quantification FL (version 2.3.3 et version 2.3.4), Deconvolution (version 1.1.7 et version 1.1.8) et FISH (version 3.2.3).L’IA a été formée par exemple pour classer les régions du tissu. Le classificateur d’apprentissage en profondeur Mininet formé à l’IA HALO a été utilisé pour classer la paroi coronaire et la graisse périvasculaire correspondante dans toutes les sections et pour éliminer l’autofluorescence de fond. La segmentation cellulaire a été réalisée en formant un classificateur de réseau de segmentation des noyaux AI avec des noyaux teints DAPI à la fois sur les images cousues de tissu coronarien entier et sur des expériences in vitro ’. La quantification des sondes était basée sur la couleur et le seuil d’intensité d’image constant, maintenant des paramètres constants à travers les échantillons. Les algorithmes d’analyse d’imagerie HALO et le flux de travail d’analyse spatiale ont été utilisés pour quantifier le nombre de points positifs par cellule et la fréquence des cellules positives divisées par les cellules totales.Le classificateur d’apprentissage profond Mininet formé par HALO AI a été utilisé pour classer la paroi coronaire et la graisse périvasculaire correspondante dans toutes les sections et pour supprimer l’autofluorescence de fond. La segmentation cellulaire a été réalisée en formant un classificateur de réseau de segmentation de noyaux d’IA avec des noyaux colorés par DAPI sur les images cousues de tissu coronaire entier et les expériences in vitro. La quantification des sondes a été basée sur le seuillage de la couleur et de l’intensité de l’image constante, en maintenant les paramètres constants entre les échantillons. Les algorithmes d’analyse d’imagerie HALO et le flux de travail d’analyse spatiale ont été utilisés pour quantifier le nombre de points positifs par cellule et la fréquence des cellules positives divisées par les cellules totales.Le classificateur d’apprentissage en profondeur Mininet formé à l’IA HALO a été utilisé pour classer la paroi coronaire et la graisse périvasculaire correspondante dans toutes les sections et pour éliminer l’autofluorescence de fond. La segmentation cellulaire a été réalisée en formant un classificateur de réseau de segmentation des noyaux AI avec des noyaux teints DAPI à la fois sur les images cousues de tissu coronarien entier et sur des expériences in vitro ’. La quantification des sondes était basée sur la couleur et le seuil d’intensité d’image constant, maintenant des paramètres constants à travers les échantillons. Les algorithmes d’analyse d’imagerie HALO et le flux de travail d’analyse spatiale ont été utilisés pour quantifier le nombre de points positifs par cellule et la fréquence des cellules positives divisées par les cellules totales.La segmentation cellulaire a été réalisée en formant un classificateur de réseau de segmentation des noyaux AI avec des noyaux teints DAPI à la fois sur les images cousues de tissu coronarien entier et sur des expériences in vitro ’. La quantification des sondes était basée sur la couleur et le seuil d’intensité d’image constant, maintenant des paramètres constants à travers les échantillons. Les algorithmes d’analyse d’imagerie HALO et le flux de travail d’analyse spatiale ont été utilisés pour quantifier le nombre de points positifs par cellule et la fréquence des cellules positives divisées par les cellules totales.La segmentation cellulaire a été réalisée en formant un classificateur de réseau de segmentation des noyaux AI avec des noyaux teints DAPI à la fois sur les images cousues de tissu coronarien entier et sur des expériences in vitro ’. La quantification des sondes était basée sur la couleur et le seuil d’intensité d’image constant, maintenant des paramètres constants à travers les échantillons. Les algorithmes d’analyse d’imagerie HALO et le flux de travail d’analyse spatiale ont été utilisés pour quantifier le nombre de points positifs par cellule et la fréquence des cellules positives divisées par les cellules totales.Les algorithmes d’analyse d’imagerie HALO et le flux de travail d’analyse spatiale ont été utilisés pour quantifier le nombre de points positifs par cellule et la fréquence des cellules positives divisées par les cellules totales.Les algorithmes d’analyse d’imagerie HALO et le flux de travail d’analyse spatiale ont été utilisés pour quantifier le nombre de points positifs par cellule et la fréquence des cellules positives divisées par les cellules totales.
IHC
H&E et IHC chromogénique ont été réalisées par la CBRD NYULH. IHC pour la quantification des macrophages a été réalisé sur une plateforme Ventana Medical Systems Discovery Ultra en utilisant du CD68 anti-humain de lapin (ref. 65) (Systèmes Médicaux Ventana). Les images ont été acquises dans la pathologie numérique multimodale Vectra Polaris. L’évaluation histopathologique et les rapports de pathologie coronarienne ont été effectués par un pathologiste cardiovasculaire clinique en aveugle. Quantification du pourcentage de CD68+ la surface a été réalisée avec le module de comptage cellulaire hybride du microscope Keyence BZ-X800. CD68+ le nombre et la fréquence des cellules ont été obtenus à l’aide d’algorithmes d’analyse par imagerie HALO et de segmentation des noyaux AI.
Immunofluorescence
Les sections coronaires d’autopsie ont été colorées avec des anticorps primaires à 4 ° C pendant la nuit et avec des anticorps secondaires à température ambiante pendant 2 h, et les noyaux cellulaires ont été colorés avec du DAPI. L’autofluorescence a été éteinte avec TrueBlack Lipofuscin Autofluorescence Quencher ( Biotium ). Les macrophages et les cellules en mousse cultivés ont été colorés avec un anticorps anti-SARS-CoV-2 NP ( ProScience ) pendant la nuit à 4 ° C, suivi d’un anti-souris caprine Alexa Fluor 488 ( Invitrogène ) pendant 2 h à température ambiante. Des images ont été acquises à l’aide d’un microscope Keyence BZ-X800. Les anticorps primaires et secondaires utilisés sont répertoriés dans le tableau supplémentaire 5.
scRNA-seq d’artères carotides humaines
Des plaques carotides ont été obtenues auprès de patients atteints de CEA (n = 10 ) inscrit à l’étude Athero-IN ( IRB 11 – 01427 et IRB i21-00429 ). Les critères d’exclusion pour l’inscription des patients étaient les suivants: infection active, maladies auto-immunes, cancer actif ou récurrent et insuffisance rénale sévère nécessitant une dialyse. Tableau supplémentaire 4 résume les caractéristiques cliniques et démographiques de la cohorte d’étude. Les plaques ont été classées par un pathologiste cardiovasculaire clinique ( N.N. ) comme fibrocalcifique (n = 3 ) et fibroathérome (n = 4 ). Trois des 10 tissus de la plaque n’ont pas été classés en raison de l’insuffisance tissulaire.
Isolement cellulaire des tissus athérosclérotiques carotides
Des échantillons de plaque fraîche ont été placés immédiatement dans du DMEM (Gibco) et traités en 30 min pour obtenir une suspension unicellulaire selon un protocole de digestion décrit précédemment66. En résumé, les échantillons ont été lavés et digérés dans du DMEM contenant 10% de FBS (Gibco, 10082147); de la collagénase de type IV (Sigma-Aldrich, C5138) à une concentration finale de 1 mg ml−1; et DNase I (Sigma-Aldrich, DN25), hyaluronidase (Sigma-Aldrich, H3506), collagénase de type XI (Sigma-Aldrich, C7657) et collagénase de type II (Sigma-Aldrich, C6885), chacun à une concentration finale de 0,3 mg ml−1 pendant 40 min dans un doux MACS Octo Dissociator ( Miltenyi Biotec ). Le tissu digéré a été filtré séquentiellement à travers des déformateurs cellulaires de 70 μm et 40 μm ( Thermo Fisher Scientific, 22363547 et 22363548 ) et centrifugé à 300g pendant 8 min. Les cellules mortes ont été prélevées avec le kit EasySep Dead Cell Removal (Annexin V) (STEMCELL Technologies, 17899), et les leucocytes ont été isolés avec le kit EasySep Release Human CD45 Positive Selection Kit (STEMCELL Technologies, 100-0105) selon les instructions du fabricant’s. Les cellules vivantes ont été comptées avec le compteur de cellules automatique Cellometer Auto 2000 (Nexcelom), et les cellules ont été chargées dans le Chromium Controller (10x Genomics). les bibliothèques ScRNA-Seq ont été préparées en utilisant le réactif Chromium Single Cell 3′ Library v3, Gel Bead et Multiplex Kit and Chip Kit (10x Genomics). La quantité et la qualité de la banque d’ADN ont été mesurées avec le test fluorométrique Qubit dsDNA HS (Qiagen) et Bioanalyzer (Agilent) et séquencées sur un séquenceur NovaSeq 6000 (Illumina). les données SCRNA-seq de six échantillons ont été précédemment publiées66 et sont disponibles dans le GEO (GSE224273). Quatre échantillons supplémentaires ont été traités pour obtenir des données scRNA-seq supplémentaires ( GEO: GSE235437).
Analyse informatique de scRNA-seq coronaire et carotidien
Cell Ranger Single-Cell Software Suite (version 3.1.0) a été utilisé pour démultiplexer et aligner à la version de référence du génome humain GRCh38. Un total de 20 639 CD45+ les cellules ont été analysées de même que les résultats de Cell Ranger des données scRNA-seq du CEA avec une moyenne de 104 351 lectures moyennes par cellule et 3 128 comptes d’identificateurs moléculaires uniques médians (UMI) par cellule. Un ensemble de données scRNA-seq (GSE131780) comprenant des échantillons coronariens athérosclérotiques humains a été obtenu à partir du référentiel de données GEO15. Au total, 12 200 cellules coronaires provenant de sept échantillons de tissus prélevés sur quatre patients ont été analysées. Les sorties de Cell Ranger avaient une moyenne de 62 328 lectures moyennes par cellule et 2 703 comptes UMI médians par cellule.
Les matrices d’expression du gène filtré de sortie ont été analysées à l’aide du package Seurat ( version 4.0.3 )67. Les gènes mitochondriaux > 10% à > 20%, < 200 gènes et < 10 000 à < 20 000 UMI ont été filtrés. Les matrices d’expression génique ont été normalisées à l’aide de la fonction SCTransform, et une analyse robuste des composants principaux a été utilisée pour l’intégration. La fonction RunPCA a été utilisée pour calculer les 30 principaux composants principaux à l’aide de gènes exprimés de manière variable. FindIntegrationAnchors a servi à identifier les ancres entre les échantillons de carotides et a été entré dans la fonction IntegrateData pour corriger les effets de lot. Les cellules ont reçu un score de cycle cellulaire en utilisant la fonction CellCycleScoring pour régresser les variations indésirables. RunUMAP avec les fonctions FindNeighbors et FindClusters a été utilisé pour le clustering cellulaire. L’intégration / co-clustering de cellules uniques carotides et coronaires a été réalisée à l’aide de Harmony68. La réduction de la dimensionnalité générée par Harmony a été utilisée pour calculer l’approximation et la projection uniformes des variétés ( UMAP ) et le regroupement basé sur des graphiques avec une résolution de 0,7. FindAllMarkers a été utilisé pour trouver des gènes exprimés différemment ( DEGs ) en utilisant le test statistique de la somme de rang de Wilcoxon, et les principales populations cellulaires ont été annotées en utilisant des DEG et des gènes marqueurs canoniques. Une analyse sous-clusternelle des cellules myéloïdes a été effectuée avec la même approche décrite ci-dessus. Package MiloR ( version 1.3.1 )55 a été utilisé pour tester l’abondance différentielle entre la carotide et la coronaire. Les quartiers cellulaires ont été définis sur un k-voisin le plus proche ( kNN ) graphique (k = 20, d = 30), et l’essai de l’abondance différentielle a été fait en utilisant un cadre de modèle linéaire général binomial négatif. Les quartiers cellulaires ont été annotés en fonction de leurs grappes, et l’abondance différentielle a été exprimée sous forme de changements de pli logarithmique (FC) affichés dans une parcelle de terrain humide.
Analyse des données GTEx
L’analyse de l’expression génique des facteurs d’entrée du SRAS-CoV-2 dans l’aorte, les artères coronaires et tibiales, le cœur (appendice auriculaire et ventricule gauche), le poumon et le sang total a été réalisée à l’aide des données GTEx (version V8, numéro d’accession dbGaP phs000424.v8.p2), qui contient 17 382 échantillons de tissus ARN-seq au total provenant de 948 donneurs décédés (67,1% d’hommes). La cohorte comprend des ascendants Caucasiens (84,6%), Afro-Américains (12,9%), Indiens d’Amérique (0,2%), Asiatiques (1,3%) et inconnus (1,1%). Les causes de décès enregistrées sont les lésions traumatiques, les maladies cérébrovasculaires (>22%) ou les maladies cardiaques (>40%). Les résultats sont présentés sous forme d’échelle logarithmique de transcrits par million (log10 TMP+1‘).
Analyse computationnelle des données scRNA-seq murines
Séquençage des données de la réf. 16 ont été extraits du numéro d’adhésion de BioProject PRJNA626450. Le contrôle de la qualité des données scRNA-seq a été effectué en utilisant FastQC (version 0.11.7). Les billes ont été alignées sur le génome de référence GRCm39 (mm39) en utilisant STAR (version 2.6.1d). FeatureCounts du paquet sous-lu (version 1.6.3) a été utilisé et les comptes normalisés ont été utilisés pour l’analyse en aval en utilisant le paquet Seurat R (version 4.3.0). Des gènes très variables ont été identifiés en utilisant la fonction FindVariableFeatures. La fonction RunUMAP avec les paramètres par défaut a été utilisée avec les fonctions FindNeighbors et FindClusters pour le clustering de cellules. L’analyse différentielle de l’expression génique a été réalisée à l’aide de la fonction FindMarkers pour identifier les différences entre les rapporteurs de Tomates monocolores (Myh11-CreERT2, Rosa26tdTomato/Tomtdato, ApoE−/−les souris ) ont nourri un régime riche en graisses ( 21% de matières grasses laitières anhydres, 19% de caséine et 0,25% de cholestérol ) pendant 18 semaines par rapport aux souris témoins. La méthode Benjamini – Hochberg a été appliquée pour contrôler le taux de fausses découvertes ( FDR ).
Expériences en biosécurité niveau 3
Des études impliquant une infection au SRAS-CoV-2 ont été approuvées par le comité institutionnel de biosécurité ( IBC21-000079 ) de la NYU Grossman School of Medicine. Toutes les procédures de niveau 3 de biosécurité ont été menées conformément au manuel de biosécurité et aux procédures opérationnelles standard de l’installation de haute maîtrise de la NYU Grossman School of Medicine.
Cellules et virus
Les cellules Vero E6 ( Collection de cultures de type américain, CRL-1586 ) ont été maintenues dans des milieux de culture DMEM contenant 10% de FBS ( Gibco ), 2 mM L-glutamine et 100 U ml− 1 pénicilline – streptomycine. Vero E6 exprimant la protéase transmembranaire, la sérine 2 et l’enzyme de conversion de l’angiotensine humaine 2 ( Vero E6-TMPRSS2-T2A-ACE2 ) ont été obtenus à partir des ressources BEI ( NR-54970 ). Les cellules Vero E6-TMPRSS2-T2A-ACE2 ont été cultivées en milieu DMEM avec 4 mM L-glutamine, 4 500 ml de glucose, 1 mM de pyruvate de sodium et 1 500 mg L− 1 bicarbonate de sodium, 10% FBS et 10 μg ml− 1 puromycine. Toutes les cellules ont été vérifiées exemptes de contamination par les mycoplasmes.
L’isolat SRAS-CoV-2 USA-WA1 / 2020 ( BEI Resources, NR52281 ) a été amplifié une fois dans les cellules Vero E6 infectées par une multiplicité d’infection ( MOI ) de 0,01 comme décrit précédemment69. Le virus a été collecté à 72 hpi lors de l’observation de l’effet cytopathique. Les débris ont été retirés par centrifugation et passage à travers un filtre de 0,22 μm, et le surnageant a ensuite été aliquoté et stocké à − 80 ° C. Le titre viral a été calculé par dosage de plaque sur les cellules de Vero E6 et informé en tant qu’unités formant des particules par millilitre ( PFU ml−1‘). Les stocks de virus ont été séquencés lors de la production de stocks viraux. Amplicon PCR couvrant le gène S (FW: gttcagagtttattctagtgcgaataattgcacttttg, RV: gcagtaaggatggctagtgtaactagcaagaataccac) a été purifié à l’aide du Nucleospin PCR and Gel Extraction Kit (Macherey-Nagel) et du Sanger séquencé (GENEWIZ) avec les amorces suivantes (FW: ggttttattgttactttcc et FW: ctaaggttaattttttc). icsARS-CoV-2 mNG virus rapporteur a été obtenu à partir de l’UTMB World Reference Center for Emerging Viruses and Arbovirus21.
Infection in vitro du SRAS-CoV-2 par des cellules primaires humaines
Les monocytes primaires humains ont été différenciés en macrophages en présence d’un facteur de stimulation des colonies de macrophages humains de 20 nM ( M-CSF, PeproTech ) dans des milieux de culture DMEM complétés par 10% FBS, 2 mM L-glutamine et 100 U ml− 1 pénicilline – streptomycine pour 5 d. Les macrophages ont ensuite été traités avec 10 μg ml− 1 de Dil-oxLDL ou ox-LDL ( Invitrogène ) pendant 1 h avant l’infection et conservé tout au long de l’expérience. Les cellules musculaires lisses aortiques humaines ( PromoCell, C-12533 ) ont été cultivées dans un milieu de croissance des cellules musculaires lisses complet avec 0,05 ml / ml FCS, 0,5 ng ml− 1 facteur de croissance épidermique humain recombinant, 0,2 ng ml− 1 facteur de croissance du fibroblast basique humain recombinant et 5 µg ml− 1 insuline humaine recombinante et traitée avec 10 µg ml− 1 Cholestérol – méthyl-β-cyclodextrine ( Sigma-Aldrich ) pendant la nuit avant l’infection par l’isolat du virus SRAS-CoV-2 USA-WA1 / 2020 au MOI 0,1 dans les milieux infectieux avec 2% de FBS. Les cellules infectées par la moquerie ont été utilisées comme témoins. Les surnageants de culture cellulaire ont été inactivés avec irradiation UV pendant 15 min et stockés à − 80 ° C. Les cellules ont été collectées dans TRIzol ( Invitrogène ) ou 10% de formol tamponné. Les macrophages et les cellules en mousse ont été traités avec du trifluoroacétate d’EG00229 ( Tocris, 6986 ) à une concentration finale de 100 μM pendant 1 h avant l’infection.
Coloration lipidique avec Oil Red O
VSMC traités avec 10 µg ml− 1 de cholestérol – méthyl-β-cyclodextrine ou véhicule pendant 48 h ont été fixés avec 10% de formol tamponné pendant 1 h et incubés avec 60% d’isopropanol pendant 1 min. Coloration avec une solution de travail O ( Sigma-Aldrich ) a été effectuée pendant 20 minutes avant la contre-tache d’hématoxyline pendant 1 min. Des images ont été acquises avec un microscope Keyence BZ-X800.
Infection ex vivo au SRAS-CoV-2 d’explosants vasculaires humains
Les spécimens de plaque carotidienne ont été décongelés, coupés en morceaux ( environ 3 × 3 mm ) et cultivés dans des milieux de culture DMEM complétés par 10% de FBS, 2 mM L-glutamine et 100 U ml− 1 pénicilline – streptomycine pendant 2 h. Les spécimens tissulaires ont été infectés par 105 PFU ml−1de l’isolat USA-WA1 / 2020 du virus SRAS-CoV-2. Les tissus non infectés ont été utilisés comme témoins. Des échantillons de milieux culturels ont été prélevés à 24 hpi, 48 hpi et 72 hpi et inactivés par irradiation UV pendant 15 min. Les spécimens tissulaires ont été fixés pendant la nuit avec 4% de paraformaldéhyde pour l’immunofluorescence ou 3% de glutaraldéhyde pour la microscopie électronique. Les tissus utilisés pour l’ARN-seq en vrac ont été stockés dans 1 ml de réactif TRIzol. Pour les expériences de blocage du PNR-1, les tissus ont été prétraités avec un agent bloquant le PNR1 ( EG00229 trifluoroacétate ) à une concentration finale de 100 μM pendant 1 h avant l’infection.
Quantification infectieuse des particules par dosage de plaque
Des dilutions décuplées de milieux de culture conditionnés ont été ajoutées aux monocouches de cellules Vero E6-TMPRSS2-T2A-ACE2 pendant 1 h et secouées doucement toutes les 15 minutes. Après incubation, les cellules ont été superposées avec 0,8% d’agarose dans du DMEM contenant 2% de FBS et incubées pendant 36 h. Après la fixation, les bouchons d’agarose ont été retirés et les plaques ont été visualisées par coloration de solution de cristal violet. Les plaques ont été comptées et les résultats ont été exprimés en PFU ml−1. Des tests de plaques utilisant des cellules Vero E6 ont été effectués avec une période d’incubation de 72 h.
Silençage du PNR1 dans les macrophages humains et les cellules en mousse
La réduction transitoire du PNR1 a été réalisée à l’aide d’un pool de génies pré-conçu siRNA FlexiTube pour le silencieux NRP1 ( Qiagen, GS8829; Hs_NRP1_8, ACGGTCATAGACACCATA; Hs_NRPAC_7, CACGCGATTCATCATP, CTCCCAGATCACATCATCCAA ) et contrôle négatif non ciblant ( Qiagen, AllsStars Negative Control siRNA ). En bref, 3 µl de Lipofectamine RNaiMAX ( Invitrogène ) et siRNA oligos 10 nM concentration finale ont été ajoutés à un volume final de 200 µl d’OptiMEM réduit le milieu sérique ( Gibco ). Le mélange a été incubé à température ambiante pendant 20 min. Les cellules plaquées ( 60 – 80% de confluence ) ont été transfectées en ajoutant des oligos/Complexes Lipofectamine RNaiMAX dans un volume final de 1 ml avec DMEM sans sérum ni antibiotiques pendant la nuit ( ~ 16 h ) avant de remplacer le milieu par un milieu DMEM complet par 10% FBS pendant 24 h. Après 24 h,la moitié des cellules ont été traitées avec 10 µg ml− 1 oxLDL pendant 2 h avant l’infection par le virus SRAS-CoV-2 USAWA1 / 2020 isole MOI 0,1 dans le milieu infectieux ( 2% FBS DMEM ) pendant 24 h. Les cellules ont été récoltées pour l’analyse de l’ARN et des protéines, et les surnageants de culture cellulaire ont été inactivés aux UV pendant 15 minutes. Les cellules ont été fixées avec 10% de formol tamponné pendant 1 h avant la coloration du NAc.
Quantification des protéines et analyse Western Blot
Les lysats de protéines, obtenus en utilisant un tampon RIPA contenant de la phosphatase et un cocktail d’inhibiteurs de protéase (100×), ont été traités pour quantifier la concentration de protéines à l’aide du Pierce BCA Protein Assay Kit. Les anticorps suivants ont été utilisés pour l’analyse par western blot: anticorps monoclonal de lapin anti-NRP1 (Cell Signaling Technology), anticorps monoclonal de souris anti-actine (Sigma-Aldrich) et anticorps secondaires anti-lapin et anti-souris conjugués HRP (ProteinSimple). Toutes les informations sur les réactifs sont répertoriées dans le Tableau supplémentaire 5. Des marqueurs de poids moléculaire et des échantillons ont été exécutés à travers le système ProteinSimple WES, et les images ont été analysées à l’aide du logiciel Compass for Simple Western (version 6.2.0).
RT–qPCR
L’ARN a été extrait à l’aide du réactif TRIzol et des kits de microprep d’ARN à base de zol direct ( Zymo Research ) ou du mini kit RNAeasy ( Qiagen ). Transcription inverse ( Kit de transcription inverse de l’ADNc à haute capacité, Biosystèmes appliqués ) a été effectuée à 37 ° C pendant 1 h, et la réaction a été arrêtée par chauffage à 95 ° C pendant 5 min et maintenez à 4 ° C. RT – qPCR a été réalisée à l’aide d’amorces spécifiques au gène ( Tableau supplémentaire 5) en utilisant le TaqMan Fast Advanced Master Mix et le système de détection Applied Biosystems QuantStudio 6 Pro. Les conditions de cycle thermique étaient de 50 ° C pendant 2 min, suivi d’une étape à 95 ° C pendant 2 min et 40 cycles à 95 ° C pendant 1 s et 60 ° C pendant 20 s. La quantification relative du gène NP SRS-CoV-2 a été calculée à l’aide du 2− ΔΔCt méthode dans la version 2.6 du logiciel de conception et d’analyse. Les FC dans l’expression des gènes ont été normalisés en gène de contrôle d’entretien ménager et logés calculésdixFC par rapport à l’échantillon témoin ( 2 h après l’inoculum viral ). Pour le silencieux NRP1 dans les expériences in vitro, contrôle de la qualité, FC dans l’expression génétique du PNR1 le gène cible a été normalisé au GAPDH gène de contrôle de l’entretien ménager. La quantification relative a été effectuée à l’aide du 2− ΔΔCt méthode et les échantillons ont été normalisés par les témoins endogènes.
RNA-seq en vrac
L’ARN des macrophages primaires et des cellules de mousse a été extrait à l’aide du réactif TRIzol et des Kits de Microprep à ARN Direct-zol suivant les instructions du fabricant. L’ARN total du tissu athérosclérotique humain a été isolé à l’aide du réactif de lyse QIAzol (Qiagen) et de l’homogénéisateur doux MACS Octo Dissociator (Miltenyi Biotec), combiné avec le nettoyage de l’ARN à l’aide du RNAeasy Mini Kit (Qiagen). Le contrôle qualité a été réalisé avec les kits Agilent RNA 6000 Nano et Pico (Agilent Technologies) en utilisant le système Bioanalyzer Agilent 2100. Pour les expériences in vitro, la préparation de la bibliothèque poly(A) a été réalisée en utilisant Illumina Stranded ARNm Preparation and Ligation (Illumina). Pour les expériences ex vivo sur plaque athéroscléreuse humaine, le kit de préparation de la bibliothèque Revelo RNA-Seq High Sensitivity a été utilisé (Tecan). Les bibliothèques ont été quantifiées à l’aide du KAPA Library Quantification Kit (Roche),regroupé à une concentration équimolaire de 2 nM et séquencé à l’aide d’un séquenceur Illumina NovaSeq 6000.
Traitement, analyse et visualisation des données ARN-seq
Le contrôle de la qualité des données ARN-seq a été effectué à l’aide de FastQC2 ( version 0.11.7 ). Les lectures séquencées brutes ont été coupées à l’aide de la version 0.20.1 ( fastp3 pour le contrôle qualité des bases et pour éliminer les adaptateurs de séquençage. Les lectures brutes ont été alignées à l’aide de la version 2.6.1d ( de STAR à l’homme combiné )Homo sapiens) assemblage de génomes GRCh38 du Consortium de référence de génome ( GCA_000001405.15 GCF_000001405.26 ) et SRAS-CoV-2 Isolat de Washington ( USA WA1 / 2020 ) génome (: MN985325.1). Le nombre d’expressions au niveau du gène a été calculé avec la fonction featureCounts dans le package Subread ( version 1.6.3; paramètres: -g gene_id -s 2 ) en utilisant les annotations du gène humain de la version 33 de GENCODE. L’expression différentielle a été effectuée à l’aide du package R DESeq2 ( version 1.30.1 ). Pour modéliser les différences d’expression génique entre les macrophages primaires et les cellules en mousse infectés par le SRAS-CoV-2, un modèle comprenant l’état de l’infection, le point de temps et le donneur en tant que variables dépendantes a été utilisé. Pour identifier les différences d’expression génique entre les macrophages infectés et les cellules en mousse infectées, un modèle comprenant le type de cellule, le point de temps et le donneur comme variables dépendantes a été utilisé. Pour analyser la variation de l’expression des gènes à travers les points de temps et l’état de l’infection dans les macrophages et les cellules en mousse, nous avons utilisé un modèle qui incorporait l’état de l’infection, le point de temps,donneur et interaction entre le statut d’infection et le point de temps en tant que variables dépendantes pour chaque type d’échantillon séparément. Les scores IFN et SRAS-CoV-2 ont été calculés comme log2 valeurs des gènes de réponse IFN et des gènes SRAS-CoV-2 comparant la réponse des cellules macrophages et moussants à 0 hpi, 2 hpi, 8 hpi, 24 hpi et 48 hpi. Données standardisées (z-les cœurs ) ont été calculés pour chaque caractéristique en soustrayant la moyenne estimée et en divisant par l’estimation s.d. Pour le clustering hiérarchique, les données ont été tracées à l’aide du package pheatmap ( version 1.0.12 ) dans R. Une analyse d’expression différentielle des échantillons de plaque athérosclérotique infectés par le SRAS-CoV-2 a été effectuée à l’aide du paquet R DESeq2 avec le point de temps et le donneur inclus comme variables dépendantes. Pour l’analyse hiérarchique des regroupements, les valeurs normalisées ont été normalisées et tracées à l’aide du paquet phéatmap ( version 1.0.12 ) dans R. P les valeurs ont été ajustées en utilisant la correction Benjamini–Hochberg et désignées comme un astérisque. L’analyse de l’enrichissement des ensembles de gènes à l’aide de Reactome Knowledgebase 2022 et Gene Ontology Biological Process 2021 des 300 meilleurs DEG a été réalisée à l’aide d’Enrichr (https://maayanlab.cloud/Enrichr/‘)70,71. Les diagrammes à barres représentent le score combiné de 10 voies les plus pertinentes ayant une signification statistique (*P < 0,05, **P < 0,01; ***P < 0,001).
Sécrétion de protéines de cytokine et de chimiokine
Un criblage de 48 cytokines et chimiokines humaines a été réalisé en utilisant des surnageants de culture inactivés aux UV en utilisant le Bio-Plex Pro Human Cytokine Screening Panel (Bio-Rad) et la plateforme Luminex 200 du Immune Monitoring Laboratory Division of Advanced Research Technologies de la NYU Grossman School of Medicine. Les données de Luminex ont été transformées; des différences statistiquement significatives ont été calculées en utilisant des données non appariées recto-verso t– tests; et P les valeurs ont été ajustées en utilisant la correction Benjamini–Hochberg. La correction empirique des lots de Bayes (Combat) a été utilisée pour supprimer les effets de lot avant de transformer les données. L’analyse statistique a été réalisée en utilisant R (version 4.0.3). Cytokines montrant le journal2FC > 0 ont été régulés à la hausse, et les cytokines montrant log2FC < 0 ont été dérégulées. Les concentrations de TGF-Invitrogen-1 et de Caspase-8 ont été mesurées par ELISA (Invitrogen) dans le surnageant de milieux de culture clarifiés, selon les instructions du fabricant.
Microscopie électronique transmission
Après 48 hpi et 72 hpi, des échantillons athérosclérotiques infectés par le SRAS-CoV-2 ex vivo ont été fixés avec 3% de glutaraldéhyde/PBS (pH 7,4) à 4 °C. Des échantillons ont été préparés pour l’évaluation au microscope électronique par la NYU Grossman School of Medicine’s Microscopy Laboratory selon les procédures opératoires standard. Les échantillons ont été examinés par microscopie électronique à transmission. Les grilles stockées ont été imagées avec un microscope électronique à transmission Talos L120C et enregistrées à l’aide d’une caméra Gatan OneView (résolution 4 K × 4 K) avec le logiciel Digital Micrograph (Gatan Microscopy Suite).
Analyse statistique
Les analyses statistiques non décrites ci-dessus ont été effectuées à l’aide de la version 9.0 de GraphPad Prism, et les détails sont inclus dans les légendes des figures. Statistiques P les valeurs ont été calculées et déclarées sur les graphiques, et P < 0,05 a été considéré comme significatif.
Résumé du rapport
De plus amples informations sur la conception de la recherche sont disponibles dans le Résumé des rapports du portefeuille Nature lié à cet article.
Disponibilité des données
les données scRNA-seq des six artères carotides humaines ont déjà été publiées et sont disponibles dans le GEO (GSE224273). Quatre plaques carotides supplémentaires ont été traitées pour obtenir des données sc-RNasseq déposées dans le GEO (GSE235437). des lésions coronariennes des données scRNA-seq ont été obtenues auprès du GEO (GSE131780). Les données de l’ARN-seq en vrac provenant des macrophages et des expériences sur les cellules en mousse et les tissus de plaque infectés par le SRAS-CoV-2 sont déposés dans le GEO (GSE235437). Les données GTEx utilisées sont la version V8, numéro d’accès dbGAP phs000424.v8.p2, et disponible sur le portail GTEx (https://gtexportal.org/home). Murine scRNA-seq d’après réf. 16 ont été extraits du numéro d’accès de BioProject PRJNA626450. Humain (Homo sapiens) l’ensemble du génome GRCh38 ( hg38 ) a été obtenu auprès du consortium de référence du génome ( GCA_000001405.15 GCF_000001405.26 ). L’isolat de SRAS-CoV-2 à Washington ( WA1 / 2020 ) génome a été obtenu à partir du référentiel NIH ( GenBank: MN985325.1). Le réactif suivant a été déposé par les Centers for Disease Control and Prevention et obtenu par BEI Resources, NIAID, NIH: SARS-Related Coronavirus 2, Isolate hCoV-19 / USA-WA1 / 2020, NR-52281. Le réactif suivant a été obtenu par BEI Resources, NIAID, NIH Cercopithecus aethiops Cellules Épithéliales du Rein Exprimant la Protéase Transmembranaire, la Sérine et l’Angiotensine Humaine-Convertissant l’Enzyme 2 (Vero E6-TMPRSS2-T2A-ACE2, NR-54970). Toutes les autres données à l’appui des résultats de cette étude sont incluses dans l’article principal et les fichiers associés. Les fichiers de données sources sont disponibles sur GitHub à l’adresse suivante https://github.com/giannarelli-lab/SARS-CoV-2-infection-triggers-pro-atherogenic-inflammatory-responses-in-human-coronary-vessels.
Disponibilité du code
Le code utilisé pour l’analyse des données est disponible sur GitHub https://github.com/giannarelli-lab/SARS-CoV-2-infection-triggers-pro-atherogenic-inflammatory-responses-in-human-coronary-vessels.